Új madár picornavírusok - Frissítés
2015.04.10. 09:00
A madár picornavírusok elemzéséről szóló összefoglaló közlemény (Boros és mtsai., 2014b) elkészülte óta számos új, madarakból azonosított picornavírus teljes genomja is leírásra került. Ebben a bejegyzésben ezért szeretném röviden bemutatni az elmúlt közel egy évben leírt új madár picornavírusokat. Új madár picornavírusnak tekintem azt a teljes genommal rendelkező vírust, amely (i) egy új nemzetség első tagja, (ii) ismert nemzetség első, madárból azonosított tagja, vagy (iii) ismert nemzetség új fajának első tagja. Az összefoglaló közleményben bemutatott 17 madarakból azonosított picornavírus fajt alkotó legalább 21 különböző genotípus (Boros és mtsai., 2014b) mellett 4 új madár picornavírus teljes genomja vált ismerté. Ezeket a vírusokat szalakótákból (Coracias garrulus): szalakóta kobuvírus 1 (Roller kobuvirus 1 - ERKV-1), vörös vércsékből (Falco tinnunculus): falcovírus A1 (Falcovirus A1 - FaV-A1), illetve házityúkokból (Gallus gallus domesticus): orivírus A1 (Orivirus A1 - OrV-A1) házityúk picornavírus 2 (chicken picornavirus 2, ChPV-2) azonosították (Pankovics és mtsai., 2015; Boros és mtsai., 2014a, 2015; Lau és mtsai., 2014). Időközben az eddig csak részleges ismert genommal rendelkező házityúk gallivírus 1 teljes genomját is meghatározták (Lau és mtsai., 2014). Az öt új madár picornavírus mindegyike besorolható az eddig is ismert 5 madár filogenetikai klaszter (madár sapelovírus, megrivírus, passerivírus, avihepatovírus és tremovírus klaszterek) egyikébe (1. kép). Ez alól talán kivételt jelenthet a szalakóta kobuvírus 1, amely filogenetikai pozíciója (1. kép), és genom felépítése (L-3-3-4) alapján beletartozhat a passerivírus klaszterbe, azonban közelebbi szekvencia-rokonságot mutat az emlősökből azonosított kobuvírus nemzetség tagjaihoz - tekintve, hogy a Kobuvirus nemzetségbe tartozik -, mint a passerivírus klaszter madár picornavírusaihoz (Pankovics és mtsai., 2015). A kérdéses vírusról bővebb leírást találhatsz az előző bejegyzésemben. Annak eldöntésére, hogy a szalakóta kobuvírus 1 egy új madár klaszter első tagja, vagy az első madarakat is fertőzni képes emlős vírus, vagy a passerivírus klaszter távoli tagja, további rokon picornavírusok leírása szükséges. Az öt madár klaszter közül csak a megrivírus és a madár sapelovírus klaszter nem bővült új tagokkal. Emellett még mindig nem írtak le a Cardiovirus leszármazási vonalba tartozó madár picornavírust. Az új madár picornavírusok részletesebb leírására a későbbiekben kerül majd sor.
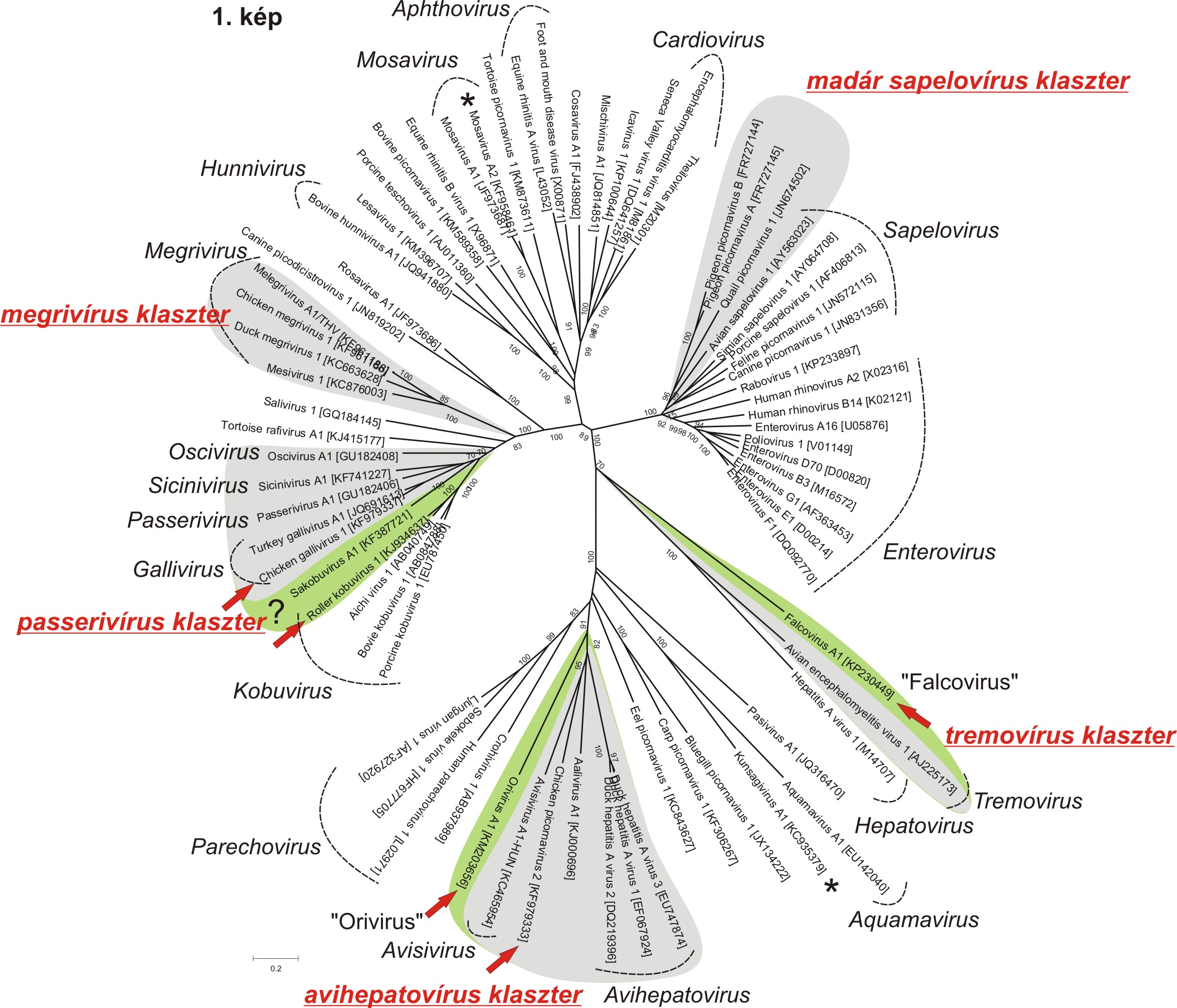
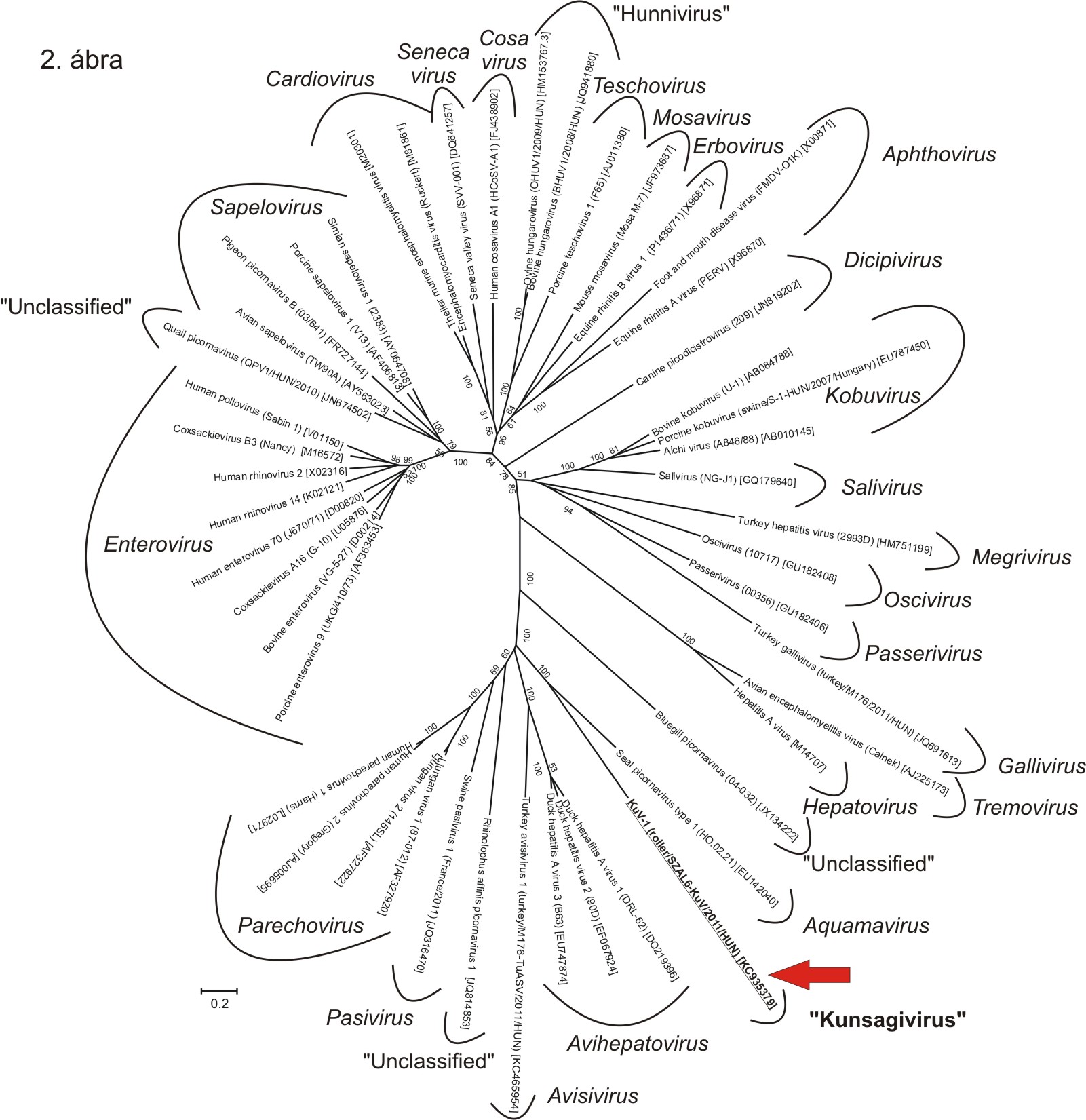
1. kép: A madár picornavírusok filogenetikai pozíciója. A hivatalos nemzetségnevek dőlt betűvel vannak szedve. Az eddig azonosított öt madár picornavírus klaszter (Boros és mtsai., 2014b) tagjait szürke háttér jelzi. Az elmúlt közel egy évben újonnan leírt madár picornavírusokat piros nyilak jelölik. Az új madár picornavírusok miatt kibővült klaszter-határokat zöld háttér jelzi. Azok a madarakból azonosított picornavírusok, amelyeknél felmerült az étrendi eredet (nem madár picornavírusok, hanem a madár által elfogyasztott prédaállat vírusai) csillaggal vannak jelölve.
Referenciák:
Boros Á, Pankovics P, Simmonds P, Pollák E, Mátics R, Phan TG, Delwart E, Reuter G. (2015). Genome analysis of a novel, highly divergent picornavirus from common kestrel (Falco tinnunculus): the first non-enteroviral picornavirus with type-I-like IRES. Infect. Genet. Evol. In press.
Pankovics P, Boros Á, Kiss T, Reuter G. (2015). Identification and complete genome analysis of kobuvirus in faecal samples of European roller (Coracias garrulus): for the first time in a bird. Arch. Virol. 160:(1) pp. 345-351.
Boros Á, Pankovics P, Adonyi Á, Phan TG, Delwart E, Reuter G. (2014a). Genome characterization of a novel chicken picornavirus distantly related to the members of the genus Avihepatovirus with a single 2A protein and a megrivirus-like 3’UTR. Infect. Genet. Evol. 28: pp. 333-338.
Boros Á, Pankovics P, Reuter G. (2014b). Avian picornaviruses: Molecular evolution, genome diversity and unusual genome features of a rapidly expanding group of viruses in birds. Infect. Genet. Evol. 28: pp. 151-166.
Lau SK, Woo PC, Yip CC, Li KS, Fan RY, Bai R, Huang Y, Chan KH, Yuen KY. (2014). Chickens host diverse picornaviruses originated from potential interspecies transmission with recombination. J Gen Virol. 95(Pt 9):1929-44.
Az első madár kobuvírus leírása - szalakóta kobuvírus 1
2015.01.30. 12:19
A Picornaviridae víruscsalád Kobuvírus nemzetségének tagjait számos emlősből kimutatták már (Reuter és mtsai., 2011). A publikus szekvencia-adatbázisokban elérhetőek az emberi (Aichi vírus), kutya, macska, egér, szarvasmarha, sertés és kecske kobuvírusok teljes szekvenciái. Emellett részleges genom szakaszok ismertek egyes denevér és patkány kobuvírusokból is. Az azonosított kobuvírusok 3 hivatalosan elfogadott (Aichivirus A-C) és egy javasolt (”Aichivirus D”) fajba sorolhatóak. Az ismert kobuvírusok mindegyike nagyon hasonló genom-felépítéssel (L-3-3-4) rendelkezik (Reuter és mtsai., 2011). A P1 genom régió előtt egy különböző hosszúságú Leader protein található, amelynek funkciója a nemzetség legtöbb tagjában ismeretlen, csak a humán Aichi vírusok Leader fehérjéjéről tudjuk, hogy fontos szerepet játszik a vírus replikációjának szabályozásában (Sasaki et al., 2003). A P1 strukturális régió három kapszid fehérjét (VP0, VP3 és VP1) kódol. A P2 és P3 nem strukturális genom régiók összesen hét fehérjét kódolnak köztük egy Hbox-NC-típusú 2A-t. A kobuvírusok legtöbbjének 5’ UTR-je V. típusú IRES-t tartalamaz. Ez alól kivételt képeznek a sertés kobuvírusok (Aichivirus C faj tagjai), amelyeknek IV-es IRES-ük van (Reuter és mtsai., 2011). Az Aichivirus A faj összes tagjának, égy a humán Aichi vírusok, valamint a kutya-, macska- és egér kobuvírusok 3’ UTR régiója tartalmazza a gallivírusoknál leírt ”súlyzó-szerű” struktúrát (részletesen lásd előző bejegyzésemet).
A kobuvírusokat még nem mutattak ki madarakból, ezért megvizsgáltuk a kobuvírusok jelenlétét RT-PCR módszerrel szalakótákban (Coracias garrulus). Az alkalmazott UNIV-Kobu-R és F általános kubuvírus szűrő primer-pár segítségével a rendelkezésünkre álló 18 kloáka minta közül háromból (17%-os pozitivitás) sikerült kobuvírust kimutatni. Az egyik kiválasztott mintában (SZAL6) található kobuvírus (SZAL6-KoV/2011/HUN) teljes genom szekvenciája (Azonosító szám: KJ934637) meghatározásra került (Pankovics és mtsai., 2015). A szalakóta kobuvírus 1 (European roller kobuvirus 1 - ERKV-1) genomja 8251 nukleotid (nt) hosszúságú, és a bevezetésben említett általános kobuvírus genom-felépítést (L-3-3-4) követi (1. kép).
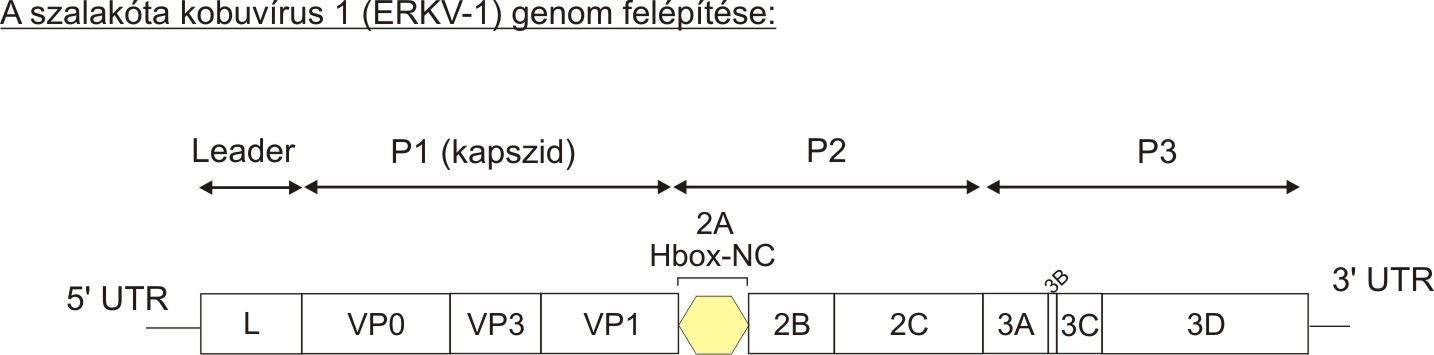
Leader proteinjének funkciója ismeretlen, 2A fehérjéje pedig a többi kobuvírus 2A-hoz hasonlóan Hbox-NC-típusú 2A fehérjék csoportjába tartozik. Törzsfejlődéstani és szekvencia-hasonlósági szempontból az ERKV-1 legközelebbi rokonságot az Aichivirus A faj tagjaival, ezen belül is a humán Aichi vírussal (72%-73%-81% aminosav egyezés a P1-P2-P3 régiókban) mutatta (2. kép).
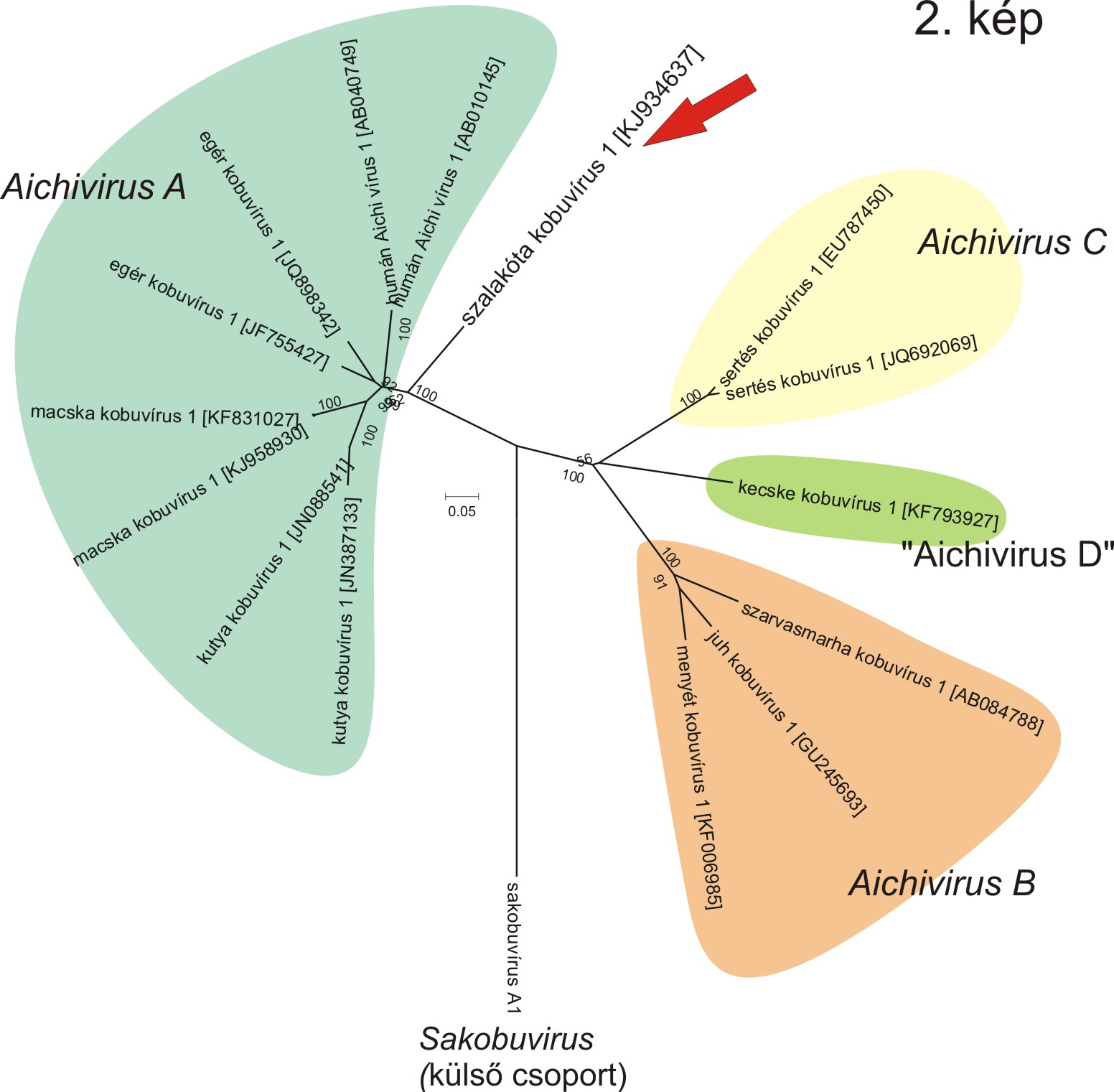
Mindezekből arra következtethetünk, hogy a szalakóta kobuvírus 1 és az Aichivirus A faj kobuvírusai azonos őstől származhatnak. A közös származásra az ERKV-1 és Aichivirus A faj tagjainak azonos, V. típusú IRES-e is utalhat. Érdekességként jelezném meg, hogy a vizsgált vírusunk 3’ UTR régiója nem tartalmazza az összes Aichivirus A faj tagjára jellemző ”súlyzó-szerű” struktúrát (Pankovics és mtsai., 2015). A filogenetikai és szekvencia-vizsgálataink eredményei nem teszik lehetővé az ERKV-1 pontos, faj-szintű besorolását. A vizsgált vírus vagy egy új kobuvírus faj (”Aichivirus E”) első tagja, vagy az Aichivirus A fajba tartozó szekvencia.
Referenciák
Sasaki J, Nagashima S, Taniguchi K. Aichi Virus Leader Protein Is Involved in Viral RNA Replication and Encapsidation. J Virol. 2003 October; 77(20): 10799–10807.
Reuter G, Boros Á , Pankovics P (2011) Kobuviruses—a comprehensive review. Rev Med Virol 21:32–41.
Pankovics P., Boros Á., Kiss T. and Reuter G. (2015). Identification and complete genome analysis of kobuvirus in faecal samples of European roller (Coracias garrulus): for the first time in bird. Arch. Virol. 60:345–351.
A súlyzó-szerű struktúra jelenléte két különböző víruscsalád tagjaiban (Picornaviridae és Caliciviridae) - új mozgó genetikai elem jelenléte RNS vírusokban?
2014.12.09. 15:37
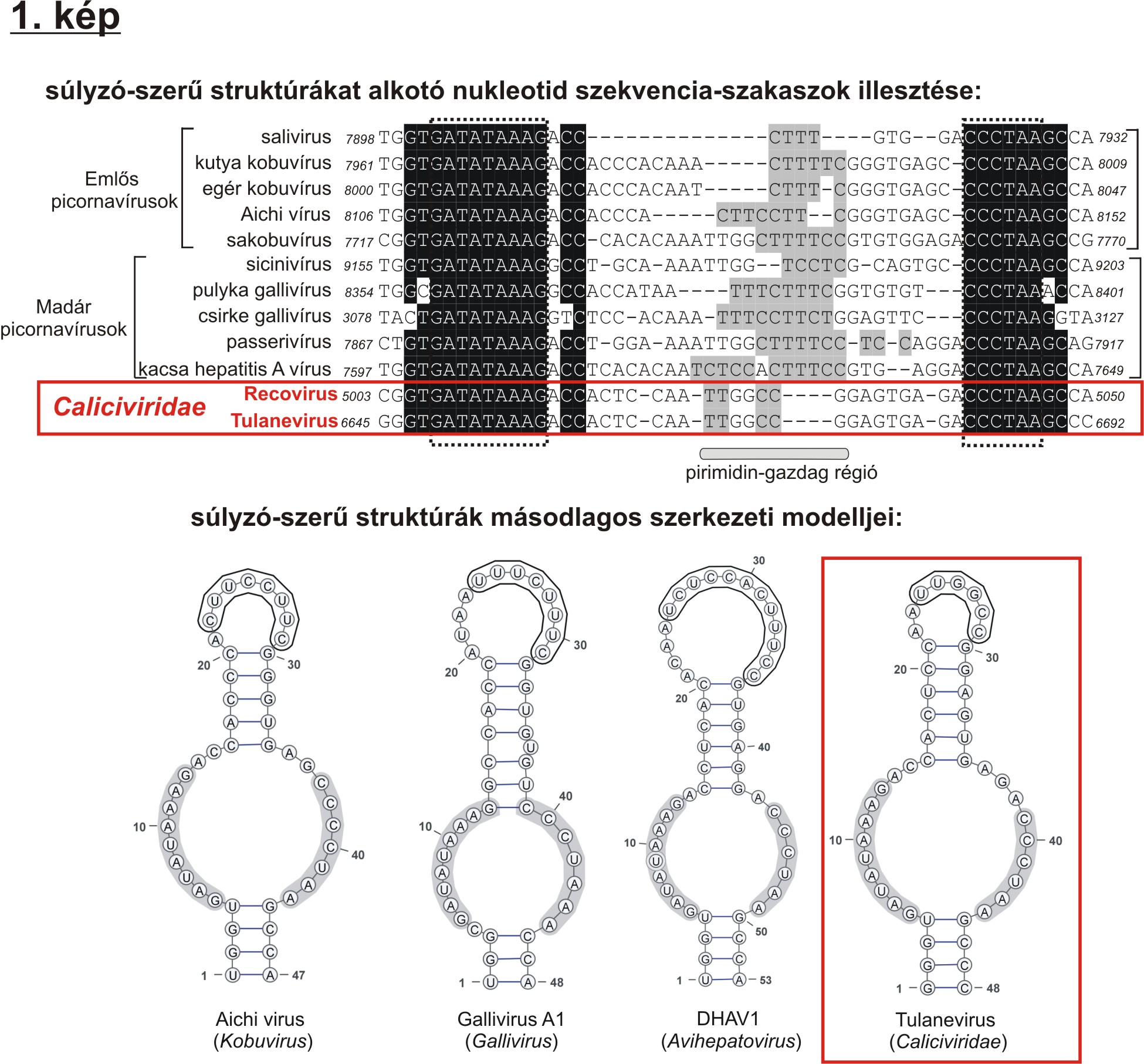
A pulyka gallivírus 3’ nem kódoló (UTR) régiójában azonosítható konzervatív, súlyzó-szerű (”barbell-like”) struktúra felépítéséről, és jelenlétéről a Picornaviridae család különböző tagjaiban a 2014.09.22.-ei, ˝Rendhagyó madár picornavírusok bemutatása 3. rész˝ című bejegyzésemben már részletesen beszámoltam. Azonban a Picornaviridae családon kívüli virális szekvencia-vizsgálatok a súlyzó-szerű struktúra jelenlétét mutatták ki a Caliciviridae család újonnan azonosított tagjaiban, a majmokból azonosított Tulane vírus (GenBank azonosító: EU391643) és az emberi hasmenéses székletből azonosított Recovírus-Bangladesh/289/2007 (GenBank azonosító: JQ745645) genomjaiban (Boros és mtsai., 2014)(1. kép). A 47 nukleotidból álló súlyzó-szerű struktúra a két calicivírus igen rövid (74 illetve 70 nukleotid hosszúságú) 3’ UTR régiójának több, mint felét alkotja. A picornavírusok súlyzó-szerű struktúrái felső hurkában található rövid pirimidin-gazdag régió, és a másodlagos RNS struktúra modelleken mindig az alsó hurok részét képező, 9+6 nukleotidból álló szekvencia-szinten is konzervatív szakasz jelenléte is kimutatható volt a két új calicivírus súlyzó-szerű struktúráiban, annyi különbséggel, hogy a felső hurok pirimidin-gazdag régiója igen rövid (1. kép). A struktúrának a virális replikációs ciklusban betöltött szerepe jelenleg még nem ismert.
A súlyzó-szerű struktúra jelenléte két különböző víruscsalád tagjaiban (Picornaviridae és Caliciviridae), valamint a struktúra STOP kodonhoz viszonyított eltérő relatív pozíciója picornavírusokban (részletesen lásd a 2014.09.22.-ei bejegyzésemet) a súlyzó-szerű struktúrának a mozgó genetikai elem funkciójára enged következtetni (Boros és mtsai., 2014).
Referencia:
Boros, Á., Pankovics, P. and Reuter, G. (2014). Avian picornaviruses: Molecular evolution, genome diversity and unusual genome features of a rapidly expanding group of viruses in birds). Infection, Genetics and Evolution 28 (2014) 151–166.
Vándormadarak picornavírusai - Kunságivírus
2014.10.30. 10:17
A jelenleg ismert madár picornavírusok döntő többségét (≈70%-át) házi szárnyasokból (kacsa, házi tyúk, pulyka, fürj) azonosították. Kutatócsoportunk ezért kezdte el vizsgálni a vadon élő madarak, ezeken belül is a vándorló madarak picornavírusait. 2013-ban sikerült egy, az addig ismert picornavírusoktól jelentősen eltérő picornavírust azonosítanunk egy jellegzetes vándormadár, a szalakóta (Coracias garrulus) székletmintájából (Boros és mtsai., 2013). A szalakóták vonuló madarak, április végén érkeznek vissza a Kárpát-medencébe afrikai telelőhelyükről és augusztus végén, szeptember elején vonul el. A kelet-európai állomány a telet Középkelet-Afrikában tölti (részletesen lásd itt). Az új picornavírust a mintavétel helyére utalva (Kiskunság tájegység) Kunsagivírus A1-nek (KuV-A1) neveztünk el. A KuV-A1 a jelenleg érvényben lévő ICTV (International Committee on Taxonomy of Viruses) Picornavírus Kutató Csoport (PSG) nemzetségbe sorolási szabályainak megfelelően (most már hivatalosan is) egy új picornavírus nemzetség (”Kunsagivirus”) alapító tagja.
A KuV-A1 genomja 7272 nukleotid (nt) hosszú a 3’ végén lévő poliA-farok nélkül. A genom egy 6747 nt (2248 aminosav/aa) hosszú nyílt leolvasási keretet (ORF) tartalmaz. A vírusgenom nem kódol N-terminális Leader fehérjét, emellett azonban a picornavírusokra jellemző felépítéssel rendelkezik: 5’ UTR-P1(VP0-3-1)-P2(2A1-2A2-B-C)-P3(3A-B-CD)-3’ UTR (1A. ábra).
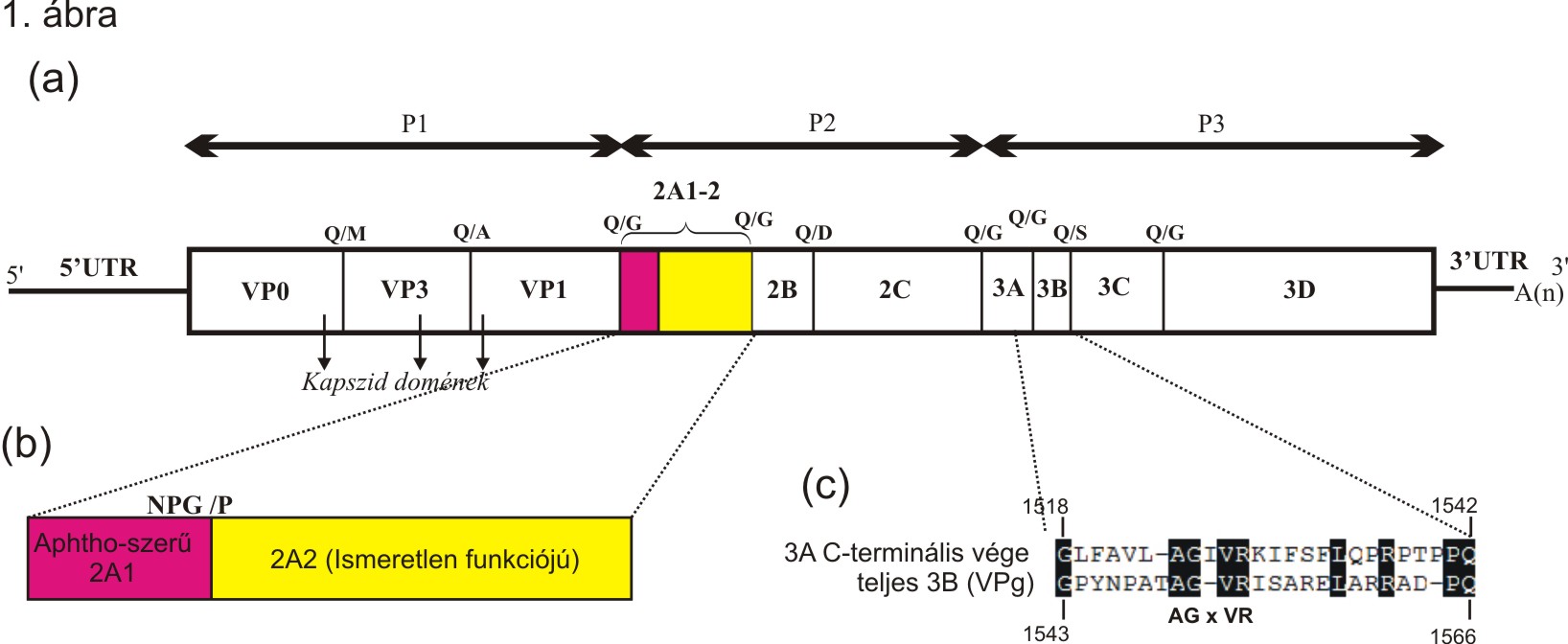
A P1, P2 és P3 régiók 26, 26 és 28%-os aminosav egyezést mutattak a legközelebbi ismert rokon, Aquamavirus nemzetségbe tartozó, 5’ UTR-P1(VP0-3-1)-P2(2A1-2A2-B-C)-P3(3A-3B1-3B2-CD)-3’ UTR genom felépítéssel rendelkező Aquamavirus A1-el (előző nevén seal picornavirus 1-el). Az aquamavírusok északi tengeri fókák (Phoca vitulina) ismert picornavírusai (Kapoor és mtsai., 2008). A rokonsági kapcsolat a kunsagivírus és az aquamavírus között a 2. ábra filogenetikai fáján is jól látható.
A KuV-A1 genomja az aquamavírusokhoz hasonlóan két 2A fehérjét (2A1 és 2A2) kódol. A 2A1 egy aphthovírus 2A-szerű, DxExNPGP-motívumot (x= bármely aminosav) (”riboszóma-ugró” hely) tartalmazó peptid. A 2A2 funkciója jelenleg ismeretlen, és csak 11%-os aminosav egyezést mutat az aquamavírusok 2A2 peptidjével (1B. ábra). Az aquamavírusoktól eltérően, ahol két 3BVPg szabadul fel a virális poliprotein hasadása folyamán, a KuV-A1 virális polipeptid hasítási helyeinek elemzése csak egy 3BVPg fehérje felszabadulására utalt. Azonban a KuV-A1 3BVPg fehérjéje kismértékű, de jelentősnek mondható hasonlóságot mutatott a virális poliproteinen szomszédosan elhelyezkedő 3A fehérje C-terminális végével (1C. ábra). A kismértékű hasonlóság mellett egy konzervatív aminosav motívum (AGxVR) is azonosítható mindkét peptiden. Mindezek arra engednek következtetni, hogy a KuV-A1-nek eredetileg, - az aquamavírusokhoz hasonlóan - két 3BVPg fehérje volt, azonban a vírus evolúciója folyamán az egyik degradálódhatott, és a 3A genom régió részévé válhatott.
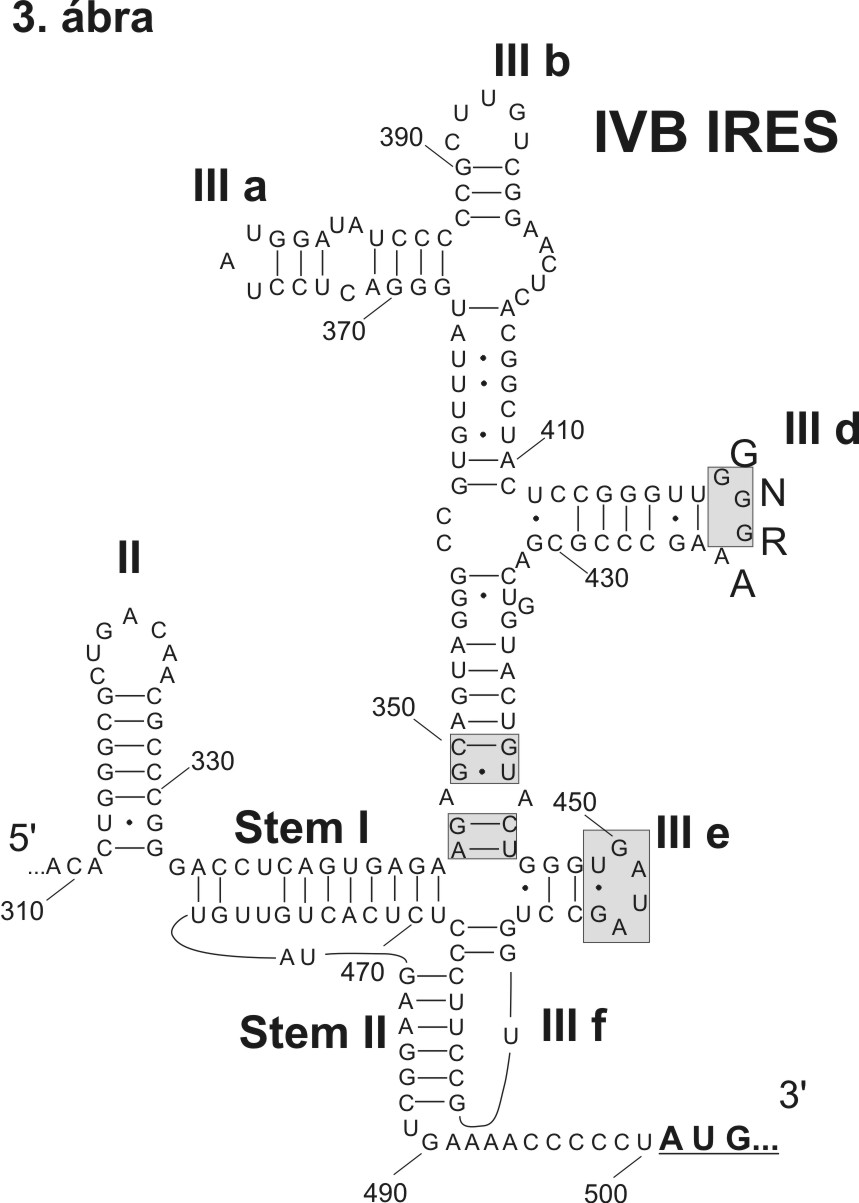
A KuV-A1 5’ UTR másodlagos RNS struktúrájának vizsgálata IVB-típusú belső riboszóma kötőhely (IRES) jelenlétét tárta fel (3. ábra). A struktúra rokonságot mutatott az aquamavírusok IRES-ével.
A KuV-A1 3’ UTR-je az ismert picornavírusok között a legrövidebb (25 nt).
A kunságivírus jelenlétét a rendelkezésünkre álló további 17 szalakóta székletminta egyikéből sem sikerült kimutatnunk, ami arra is utalhat, hogy a vizsgált vírusnak nem a szalakóta a természetes gazdaállata, hanem egy olyan állat, feltehetően kisemlős, amelyet a vizsgált madár elfogyasztott. További vizsgálatok szükségesek a KuV-A1 gazdaállat-spektrumának a feltárásához.
Referenciák
Boros, Á., Kiss, T., Kiss, O., Pankovics, P., Kapusinszky, B., Delwart, E. and Reuter, G. (2013). Genetic characterization of a novel picornavirus distantly related to the marine mammal-infecting aquamaviruses in a long-distance migrant bird species, European Roller (Coracias garrulus). J. Gen. Virol. 94: 2029–2035
Kapoor, A., Victoria, J., Simmonds, P., Wang, C., Shafer, R.W., Nims, R., Nielsen, O. and Delwart, E. (2008). A highly divergent picornavirus in a marine mammal. J. Virol. 82: 311-320.
Rendhagyó madár picornavírusok bemutatása 3. rész
2014.09.22. 15:24
Farkas és munkatársai 2012-ben több, az énekesmadarakból azonosított turdivírusokkal (jelenleg passeri-, és oscivírusok), illetve az emlősökből azonosított kobuvírusokkal (Kobuvirus nemzetség) távoli rokon picornavírust azonosítottak pulyka és csirke székletmintákból, amelyeket gallivírusként neveztek el (Farkas és mtsai., 2012). Konvencionális RT-PCR módszereket alkalmazva csak részleges, a vírusgenom P3 régiójából származó szekvenciákat határoztak meg és tettek elérhetővé a GenBankban (JF424824-30). Kutatócsoportunknak magyarországi pulykamintákból virális metagenomikai és RT-PCR módszerekkel, még szintén 2012-ben sikerült a pulyka gallivírus teljes RNS genomjának meghatározása (Boros és mtsai., 2012). A pulyka gallivírus genomja 8496 nukleotid (nt) hosszú a 3’ végén lévő poliA-farok nélkül. A genom egy 7425 nt (2474 aminosav/aa) hosszú nyílt leolvasási keretet (ORF) tartalmaz. A vírusgenom egy N-terminális Leader fehérjével illetve a picornavírusokra jellemző felépítéssel rendelkezik: 5’ UTR-L-P1(VP0-3-1)-P2(2A-B-C)-P3(3A-B-CD)-3’ UTR (1. ábra).
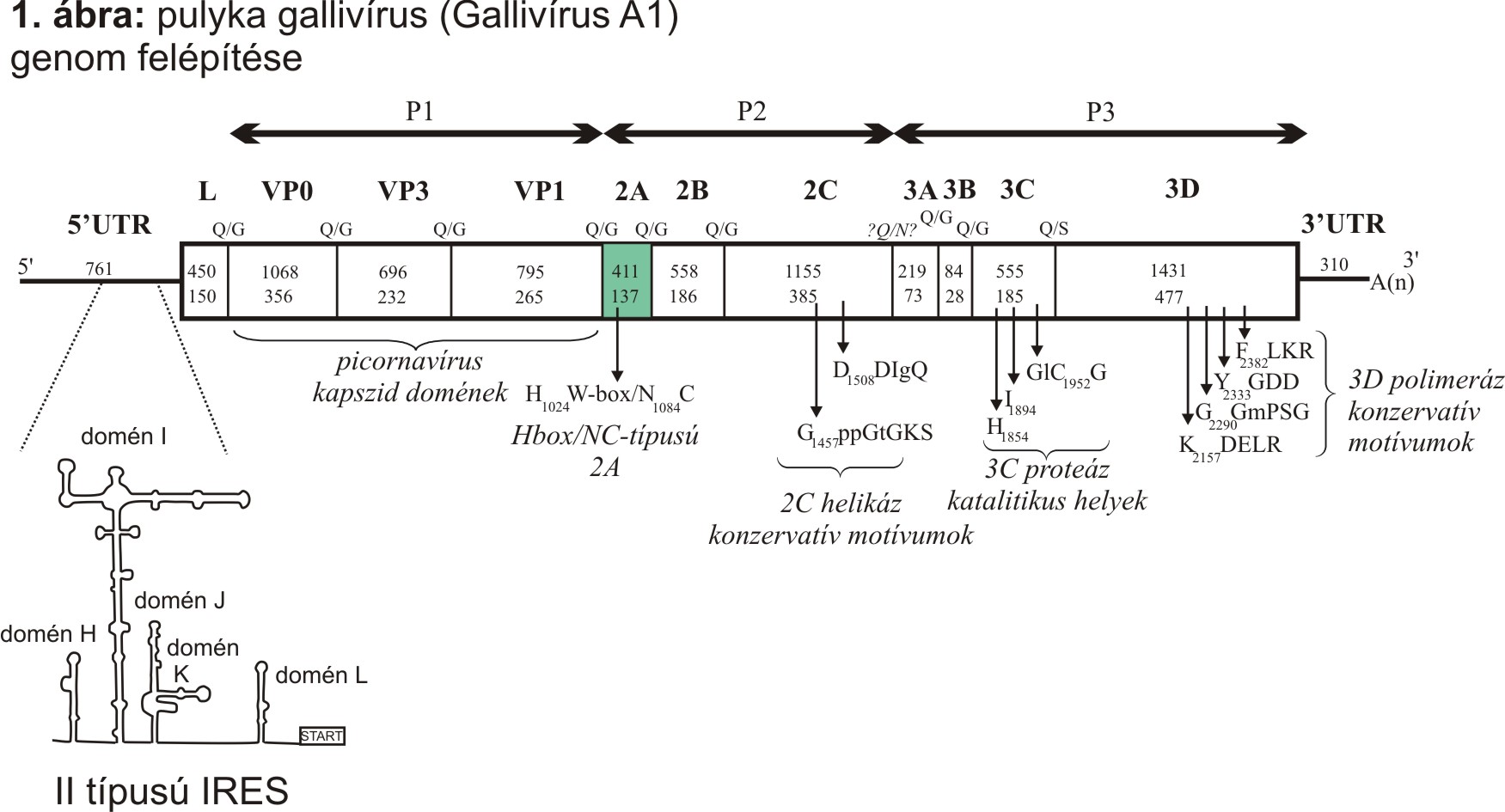
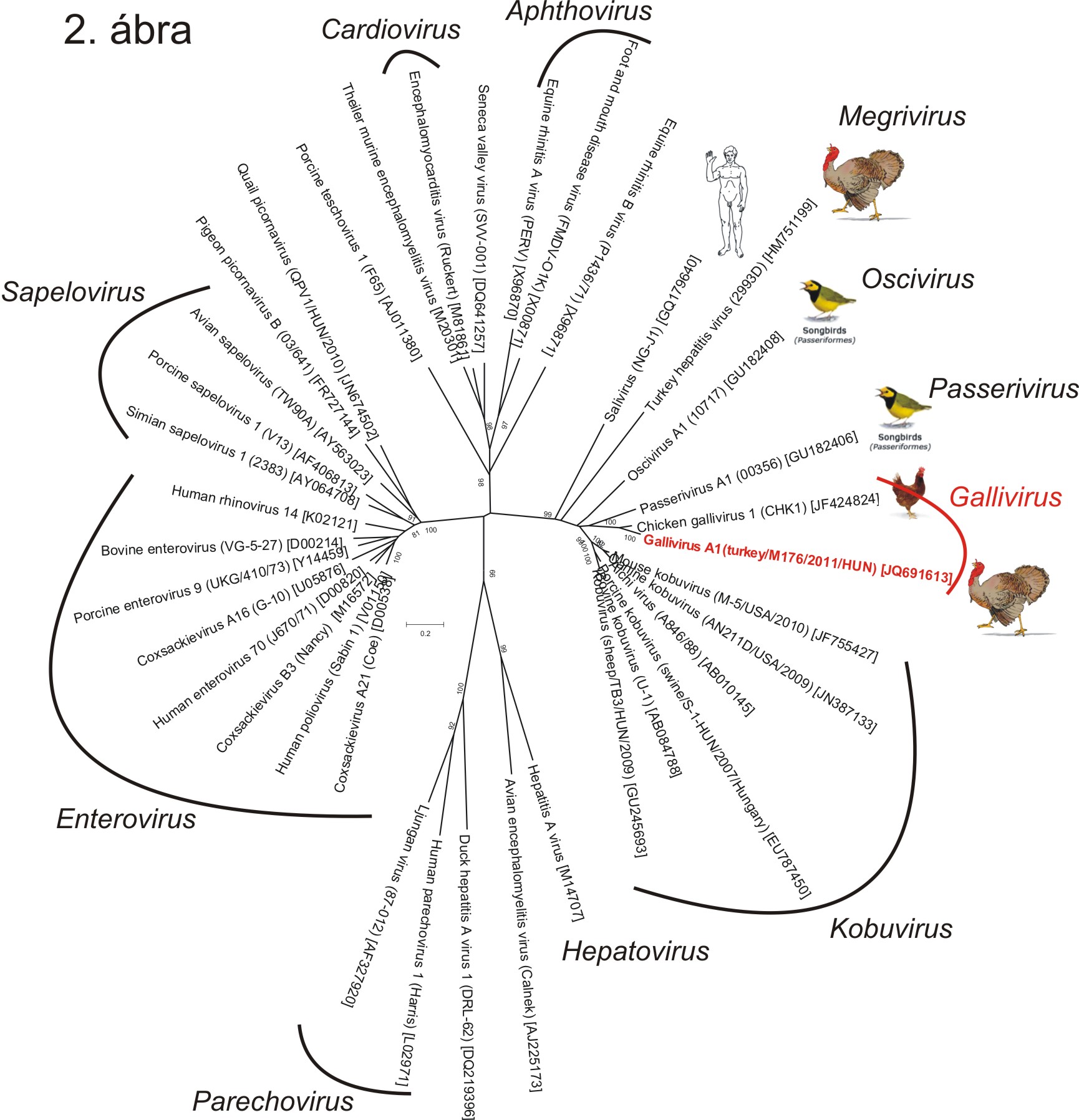
A P1, P2 és P3 régiók 18, 27/32 és 40/45%-os aminosav egyezést mutattak a legközelebbi ismert rokon, Kobuvirus nemzetségbe tartozó sertés kobuvírus 1-el (PoKV-1), illetve a Turdivirus 1-el (jelenleg a Passerivirus nemzetségbe tartozó Passerivirus A1). A vírusgenom egy 150 aa hosszúságú ismeretlen funkciójú Leader (L) proteint, illetve csak egy 137 aa hosszúságú 2A peptidet kódol, amely a Hbox/NC-típusú 2A csoportba tartozik (részletekért lásd: 2014.03.28-ai bejegyzésemet). A pulyka gallivírus távoli rokonságot mutat az énekesmadarakból azonosított passerivírusokkal és oscivírusokkal valamint az emlősök kobuvírusaival (2. ábra). A távoli filogenetikai rokonság többek közt a konzervatív P3 genom régióban, valamint az alacsony szekvencia-egyezési értékek alapján a pulyka galllivírust 2013-ban egy új nemzetségbe, a Gallivirus nemzetségbe sorolták, amelynek prototípus vírustörzse a kutatócsoportunk által azonosított pulyka gallivírus 1, újabb nevén Gallivirus A1 (GenBank ID: JQ691613) lett.
A vírus további érdekessége, hogy ez volt az első ismert madár picornavírus, amelynek 5’ UTR régiója II-es típusú belső riboszóma-kötőhelyet (Internal Ribosomal Entry Site - IRES) tartalmazott (1. ábra). II-es típusú IRES-t addig csak a filogenetikailag távoli Cardio-, Erbo-, Aphtho-, és Parechovirus nemzetségbe tartozó picornavírusok esetében írtak le (részletekért lásd: 2014.05.12-ei bejegyzésemet).
A pulyka gallivírus 310 nt hosszú 3’ UTR régiójában azonosítottunk egy 48 nt hosszúságú szakaszt, ami a másodlagos RNS struktúra modelljeink mindegyikén súlyzó alakot vett fel, és amit alakja miatt súlyzó-szerű (”barbell-like”) stuktúrának neveztük el (3. ábra). A súlyzó-szerű struktúra felső régiója egy rövid pirimidin-gazdag (C és U bázisokból álló) szakaszt tartalmazott (3. ábra).
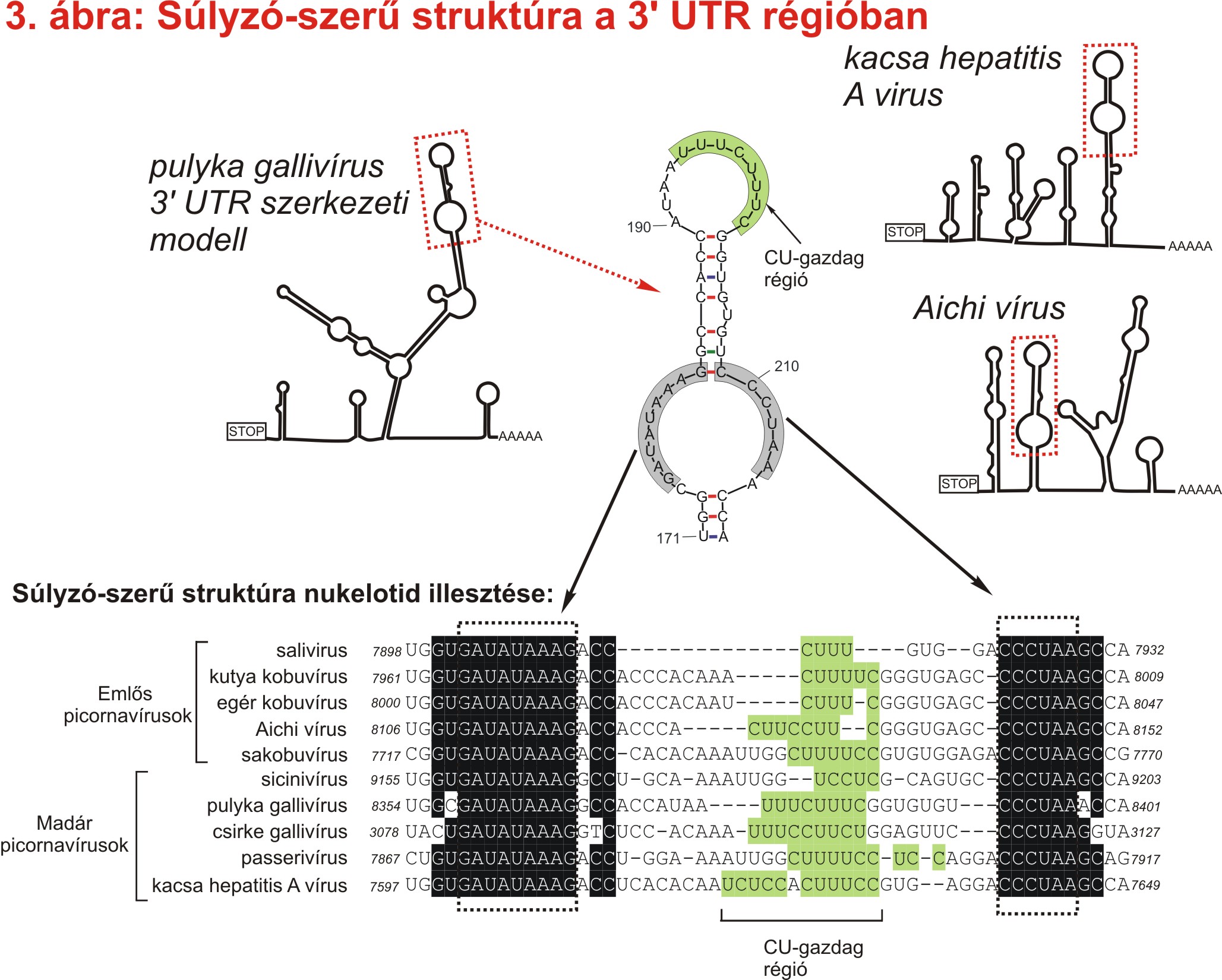
Az összes ismert picornavírusra kiterjedő szekvencia-vizsgálataink kimutatták a súlyzó-szerű struktúra jelenlétét több, a gallivírussal rokon picornavírus 3’ UTR régiójában, valamint a filogenetikailag távoli Avihepatovirus nemzetség vírusaiban is (4. ábra).
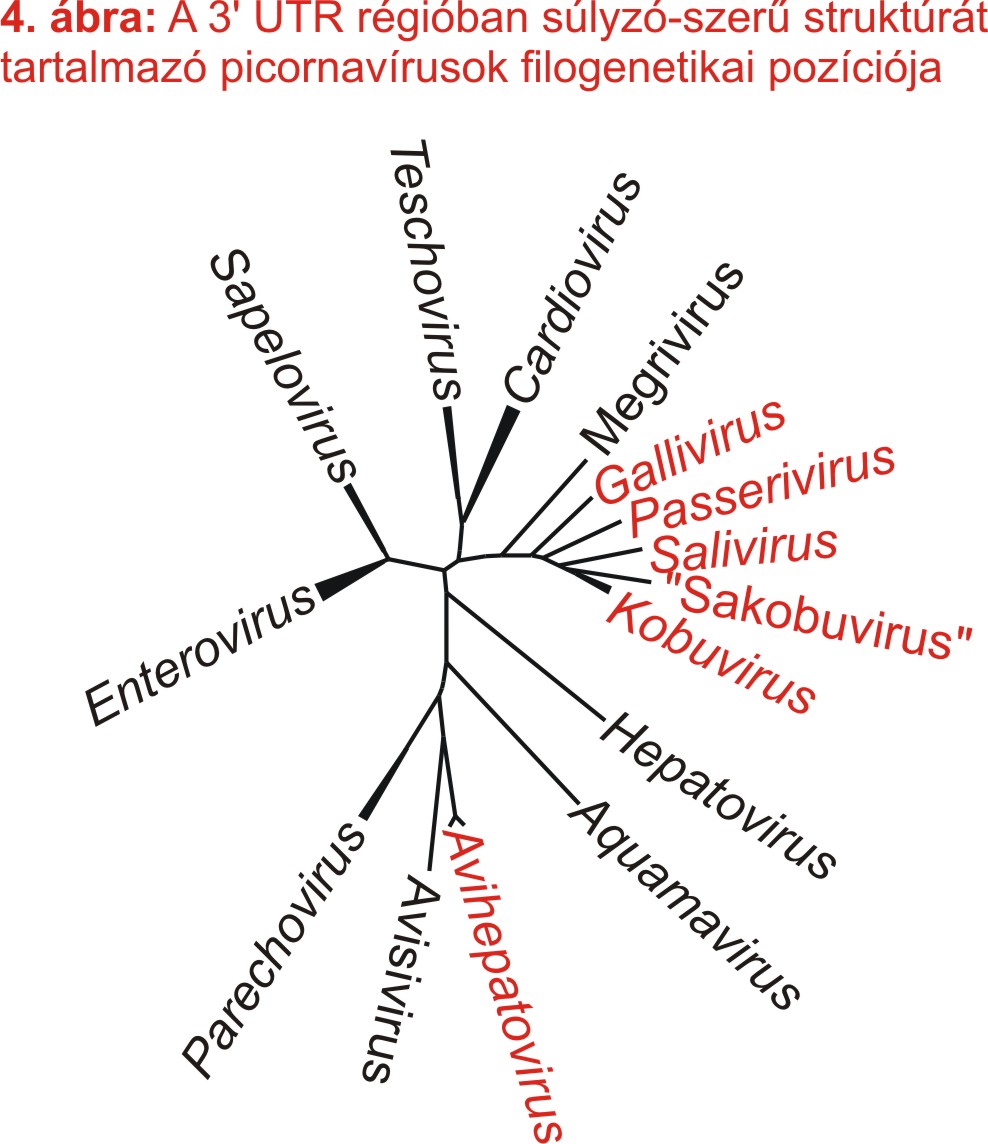
A súlyzó-szerű struktúrák a különböző picornavírusok 3’ UTR régiójából készült másodlagos RNS struktúra modelleken a STOP kodontól vírusonként eltérő távolságban helyezkedtek el (3. ábra felső része). A súlyzó-szerű struktúrákat tartalmazó 3’UTR régiók illesztése a felső hurokban található rövid pirimidin-gazdag régió, és a másodlagos RNS struktúra modelleken mindig az alsó hurok részét ábraező, 9+6 nukleotidból álló szekvencia-szinten is konzervatív szakasz jelenlétét mutatta ki (3. ábra). A súlyzó-szerű struktúrák jelenléte filogenetikailag távoli picornavírusok 3’ UTR régióiban, valamint e struktúrák eltérő relatív pozíciója együttesen a súlyzó-szerű struktúrának, mint mobil genetikai elemnek a funkciójára utalhatnak (Boros és mtsai., 2012).
A pulyka gallivírus 1 teljes genomjának leírását követően idén a csirke gallivírus teljes genom szekvenciája is meghatározásra került. A csirke gallivírus szintén II-es típusú IRES-t és a 3’ UTR-jében súlyzó-szerű struktúrát is tartalmaz (Lau és mtsai., 2014).
Referenciák
Farkas, T., Fey, B., Hargitt 3rd, E., Parcells, M., Ladman, B., Murgia, M., Saif, Y., 2012. Molecular detection of novel picornaviruses in chickens and turkeys. Virus Genes 44, 262-272.
Boros, Á., Nemes, C., Pankovics, P., Kapusinszky, B., Delwart, E., Reuter, G., 2012. Identification and complete genome characterization of a novel picornavirus in turkey (Meleagris gallopavo). J. Gen. Virol. 93, 2171–2182.
Lau, S.K., Woo, P.C., Yip, C.C., Li, K.S., Fan, R.Y., Bai, R., Huang, Y., Chan, K.H. and Yuen, K.Y. (2014). Chickens host diverse picornaviruses originated from potential interspecies transmission with recombination. J Gen Virol. 2014 Jun 6. pii: vir.0.066597-0. doi: 10.1099/vir.0.066597-0. [Epub ahead of print] PubMed PMID: 24906980.
Rendhagyó madár picornavírusok bemutatása 2. rész
2014.08.15. 12:55
Az Avisivirus nemzetséget 2013-ban hozták létre. Jelenleg egy fajból (Avisivirus A) áll, amelynek tagjait mindeddig csak madarakból (pulyka és csirke) azonosítottak. A nemzetség prototípus vírusa az Avisivirus A1 (AsV-A1, korábbi elnevezése: pulyka avisivirus 1), melyet először kutatócsoportunk azonosított 2013-ban magyarországi háziasított pulyka (Meleagris gallopavo f. domestica) mintákból (Boros és mtsai., 2013). Az AsV-A1-HUN RNS genomja 7532 nukleotid (nt) hosszúságú, amely egy 6720 nt (2239 aminosav) hosszú nyílt leolvasási keretet (ORF) tartalmaz. A vírusgenom a picornavírusokra jellemző felépítéssel rendelkezik: 5’ UTR-P1(VP0-3-1)-P2(2A-B-C)-P3(3A-B-CD)-3’ UTR (1. A. kép). 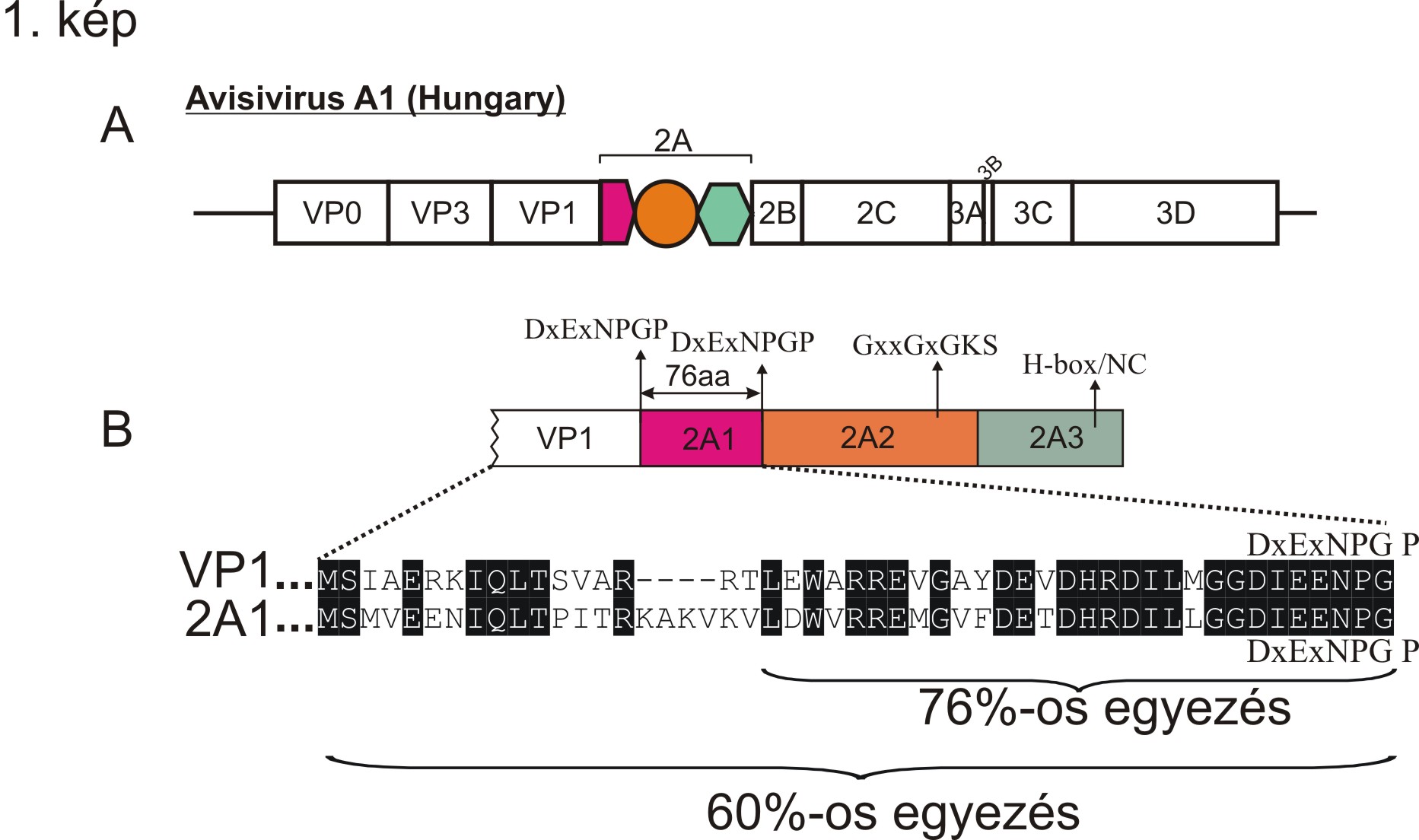
A P1, P2 és P3 régiók 33, 32 és 36%-os hasonlóságot mutattak a legközelebbi ismert rokon, Avihepatovirus nemzetségbe tartozó kacsa hepatitisz A vírusokkal (Duck hepatitis A virus - DHAV). A vírusgenom egyik legérdekesebb része a 2A genom-régió, amely a DHAV-hoz hasonlóan három, egymástól különböző peptidet (2A1-2A2-2A3) kódol (1. kép). A 76 aminosav (aa) hosszú 2A1 egy DxExNPGP motívumra (x= bármely aminosav) végződő, Aphthovírus 2A-szerű peptid. Részletesebb leírást az NPGP-tartalmú 2A-ról lásd a 2014.03.28-ai bejegyzésemet. The 140 aa hosszú 2A2 a DHAV 2A2 peptidhez hasonlóan tartalmaz egy GxxGxGKS konzervatív motívumot, amely egyben P-hurok-tartalmú nukleozid trifoszfát hidroláz enzimek katalitikus motívuma. A 123 aa hosszúságú 2A3 a DHAV 2A3-jához hasonlóan egy Hbox/NC-tartalmú 2A fehérje (1. kép). A Hbox/NC-tartalmú 2A-ról részletesebben lásd a 2014.03.28-ai bejegyzésemet. A hasonló 2A-szerkezet az avisivírusok és a DHAV-ok rokonságára utalnak, ami a 3D fehérjeszekvenciákból készült filogenetikai fán is jól látható (2. kép).
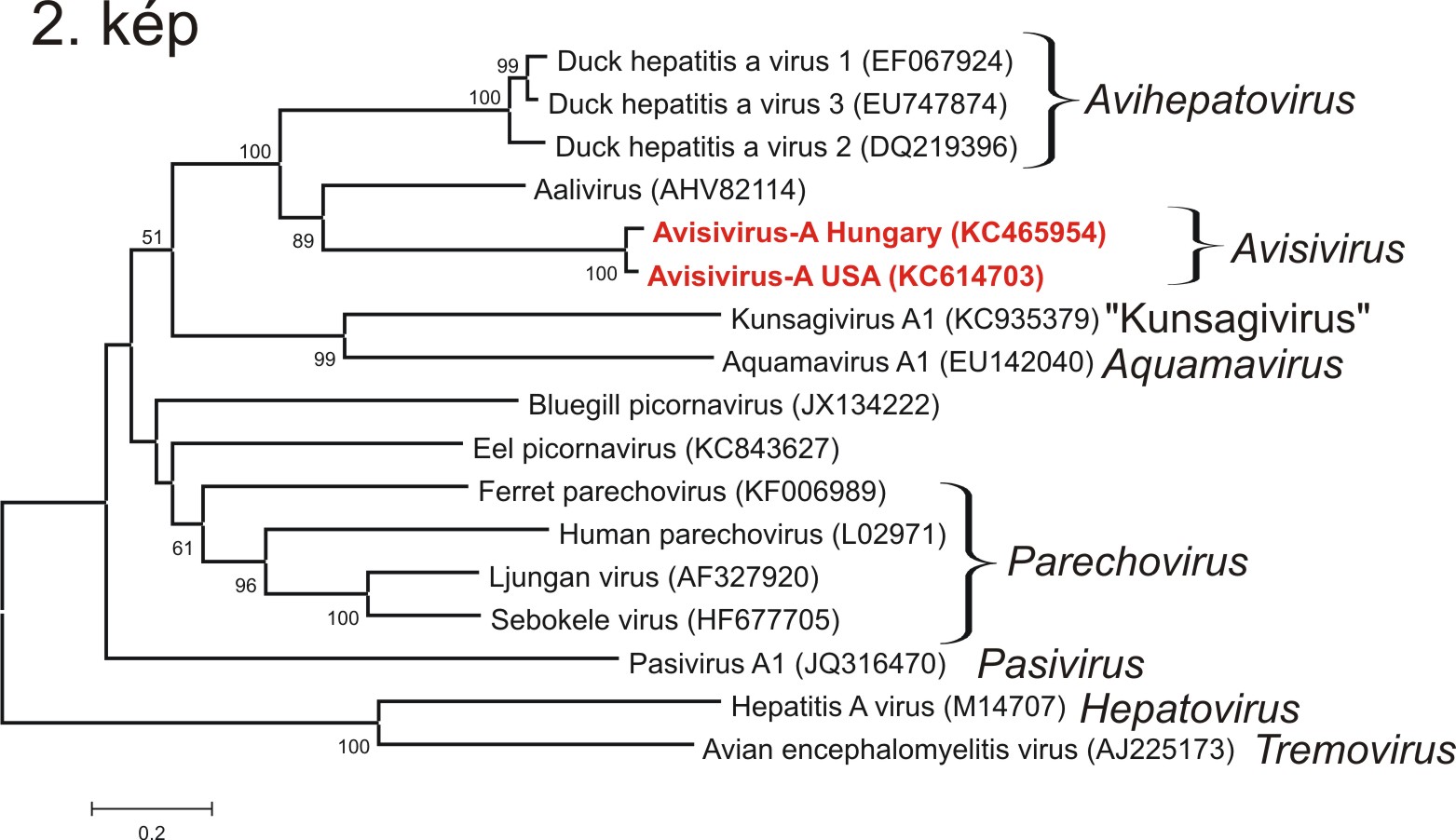
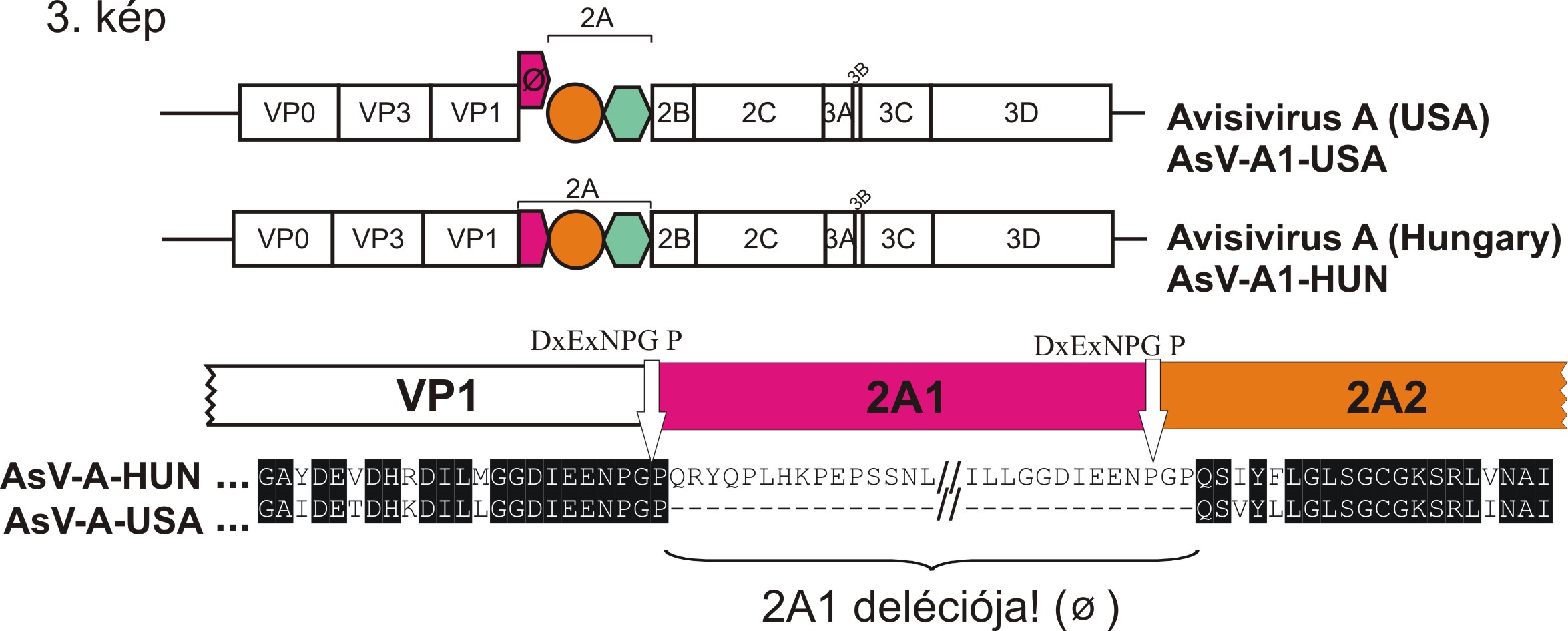
A VP1/2A régió vizsgálata egy további DxExNPGP motívum jelenlétét mutatta ki, ami a 2A1 peptid előtt helyezkedik el, és valószínűsíthető, hogy a motívum a VP1/2A hasítási helyként funkcionál. A két DxExNPGP motívum jelenléte valószínűleg egy ”génduplikáció” eredménye. Erre utal a két motívumot tartalmazó genom-szakaszok jelentős szekvencia-hasonlósága (1. B. kép). A víruscsoport első képviselőjének leírása után az avisivírus jelenlétét kimutatták az Egyesült Államokban is (AsV-A1-USA), pulykamintákból (Ng és mtsai., 2013). Az AsV-A1-USA filogenetikailag is nagyon közel állt a magyar avisivírus törzshöz (2. kép), valamint RNS genomja is hasonló felépítést mutatott az AsV-A1-HUN genomjához (3. kép). A legjelentősebb különbség a két vírustörzs között az volt, hogy az AsV-A1-USA genomja csak egy DxExNPGP motívumot tartalmazott (3. kép). A két avisivírus szekvencia illesztése alapján valószínűsíthető hogy az amerikai avisivírus törzsből hiányzik a 2A1 genom régió. Az AsV-A1-USA genomjának DxExNPGP motívuma valószínűleg a VP1 C-terminális végén helyezkedik el (3. kép). A 2A genom régió különbözősége a magyar és az amerikai avisivírus törzsekben a 2A genom régiónak, a többi genom régióhoz mérten gyorsabb evolúciós változékonyságára utalnak. Az avisivírusok jelenlétét nemrég kimutatták házi csirkékből is (Lau és mtsai., 2014), ahol magyar és amerikai avisivírusokhoz hasonlóan, a két azonosított csirke avisivírus közül az egyik (chicken picornavirus 2) két darab, míg a másik (chicken picornavirus 3) csak egy DxExNPGP motívumot tartalmazott.
Referenciák
Boros, Á., Nemes, C., Pankovics, P., Kapusinszky, B., Delwart, E., Reuter, G., 2013.Genetic characterization of a novel picornavirus in turkeys (Meleagris gallopavo) distinct from turkey galliviruses and megriviruses and distantly related to the members of the genus Avihepatovirus. J. Gen. Virol. 94, 1496–1509.
Ng. T.F., Cheung, A.K., Wong, W., Lager, K.M., Kondov, N.O., Cha, Y., Murphy, D.A., Pogranichniy, R.M., Delwart, E., 2013. Divergent picornavirus from a Turkey with gastrointestinal disease. Genome Announc. 2, e00134-13.
Lau, S.K., Woo, P.C., Yip, C.C., Li, K.S., Fan, R.Y., Bai, R., Huang, Y., Chan, K.H. and Yuen, K.Y. (2014). Chickens host diverse picornaviruses originated from potential interspecies transmission with recombination. J Gen Virol. 2014 Jun 6. pii: vir.0.066597-0. doi: 10.1099/vir.0.066597-0. [Epub ahead of print] PubMed PMID: 24906980.
Rendhagyó madár picornavírusok bemutatása 1. rész
2014.07.29. 14:46
A Megrivírus nemzetség jelenleg 4 fajból áll. A nemzetség prototípus törzsét már 1959-ben izolálták hepatitiszes tüneteket mutató háziasított pulykákból (Meleagris gallopavo f. domestica). Az izolált vírus törzset pulyka hepatitisz vírus 1-esnek (Turkey hepatitis virus 1 / THV-1) nevezték el (Snoeyenbos és mtsai., 1959). A vírus fiziko-kémiai tulajdonságai régóta ismertek, azonban RNS örökítőanyagának kódoló szekvenciáját csak 2011-ben sikerült meghatározni (Honkavuori és mtsai., 2011), az első teljes THV-1 szekvencia pedig csak 2014-ben került meghatározásra (Boros és mtsai., 2014). A THV-1 kódoló szakaszának genetikai vizsgálata kimutatta egy 1800 nukleotid hosszú, ismeretlen fehérjét/fehérjéket kódoló szakasz jelenlétét a genom VP1 és 2B régiója között, amely genom szakasz megfeletethető a picornavírusok 2A régiójának. A szekvencia analízis kimutatta egy már ismert 2A fehérje, az úgynevezett Hbox/NC típusú 2A (erről a 2A típusról részletesen olvashatsz korábbi bejegyzéseimben) jelenlétét ezen a szakaszon. Hasítási helyek elemzése további két potenciális vágási helyet azonosított, ami arra utalt, hogy a THV-1 RNS genomja legalább 3 különböző 2A fehérjét kódol (Honkavuori és mtsai., 2011). Korábban nem volt ismert hasonló picornavírus, amely ilyen hosszú 2A régióval rendelkezett volna. Virális metagenomikai és RT-PCR módszerek felhasználásával egy filogenetikailag és nukleotid-szekvenciában a THV-1-hez közeli rokon picornavírust sikerült izolálni csirkékből (Gallus gallus domesticus) (Boros és mtsai., 2014). A filogenetikai és szekvencia hasonlóság alapján a picornavírus csirke megrivírus 1 néven Megrivírus nemzetségbe sorolható. A csirke megrivírus RNS genomja a THV-1-hez hasonlóan tartalmazott egy hosszú 2A régiót, ami azonban 231 nukleotiddal rövidebbnek, mint a THV-1 azonos szakasza. A 2A régió nukleotid szekvenciáinak illesztése egy 231 nukleotid hosszú inzerciót mutatott ki a THV-1-nél, amely hiányzott a csirke megrivírus genomjából. Az inzerció további vizsgálata egy eddig még nem azonosított, potenciális hasítási hely jelenlétére utalt a THV-1 esetében. Ezek az adatok arra utalnak, hogy a THV-1 összesen 4 különböző 2A fehérjét (2A0-2A1-2A2-2A3) kódol, amelyek közül egy (2A0) valószínűleg kivágódott a csirke megrivírus evolúciós fejlődése során, amely így csak három 2A fehérjét kódol (1. kép A része). Eddigi ismereteink szerint nem ismerünk olyan picornavírust, amely ilyen nagyszámú 2A fehérjét kódolna (Boros és mtsai., 2014).Utóbbi állítás a cikk megjelenése óta hamisnak bizonyult, ugyanis leírtak egy kacsa picornavírust (”Aalivirus”) amely összesen 6 (!) 2A fehérjét kódol (Wang és mtsai., 2014).
További THV-1 rokon vírusokat is leírtak galambokból (Mesivírus 1 és 2). A mesivírusok ”különlegessége”, hogy 2A régiójuk ”csak” két különböző 2A fehérjét kódol (Phan és mtsai., 2013).
A THV-1 és a csirke megrivírusok RNS genomjának másik érdekessége a rendkívül hosszú (>600 nukleotid) 3’ nem kódoló szakasz (3’ UnTranslated Region / 3’ UTR). Szekvencia vizsgálatok egy potenciális második nyílt leolvasási keret (Open Reading Frame / ORF) jelenlétét mutatták ki közvetlenül a virális poliprotein-kódoló régió STOP kodonja után. A potenciális második ORF egy 98 (THV-1), illetve egy 114 (csirke megrivírusok) aminosav hosszú fehérjét kódolhat (Farkas és mtsai., 2012; Boros és mtsai., 2014). Az ORF2 fehérjéi csak kismértékű szekvencia-egyezést mutatnak. A második ORF teljesen hiányzik a galamb mesivírusok genomjából (1. kép B része). Ez a genom-felépítés egyedülálló az ismert picornavírusok között.
A második ORF utáni szakasz, a tényleges 3’ UTR komplex felépítéssel rendelkezik: közvetlen a második ORF után következik egy konzervatív szekvencia szakasz (Unit A), amely a THV-1-nél és a galamb mesivírusoknál ismétlődik, míg a csirke megrivírusoknál csak egy példányban van jelen (1. kép B és C része). A Unit A után egy úgynevezett AUG (Adenin, Uracil és Guanin) gazdag régió következik, amely szintén tartalmaz egy konzervatív szekvencia-szakaszt (Unit B). A Unit B egy példányban van jelen a THV-1-nél és a galamb mesivírusoknál, azonban ismétlődik a csirke megrivírusoknál (1. kép B része) (Boros és mtsai., 2014). Hasonló 3’ UTR felépítést még nem írtak le korábban.
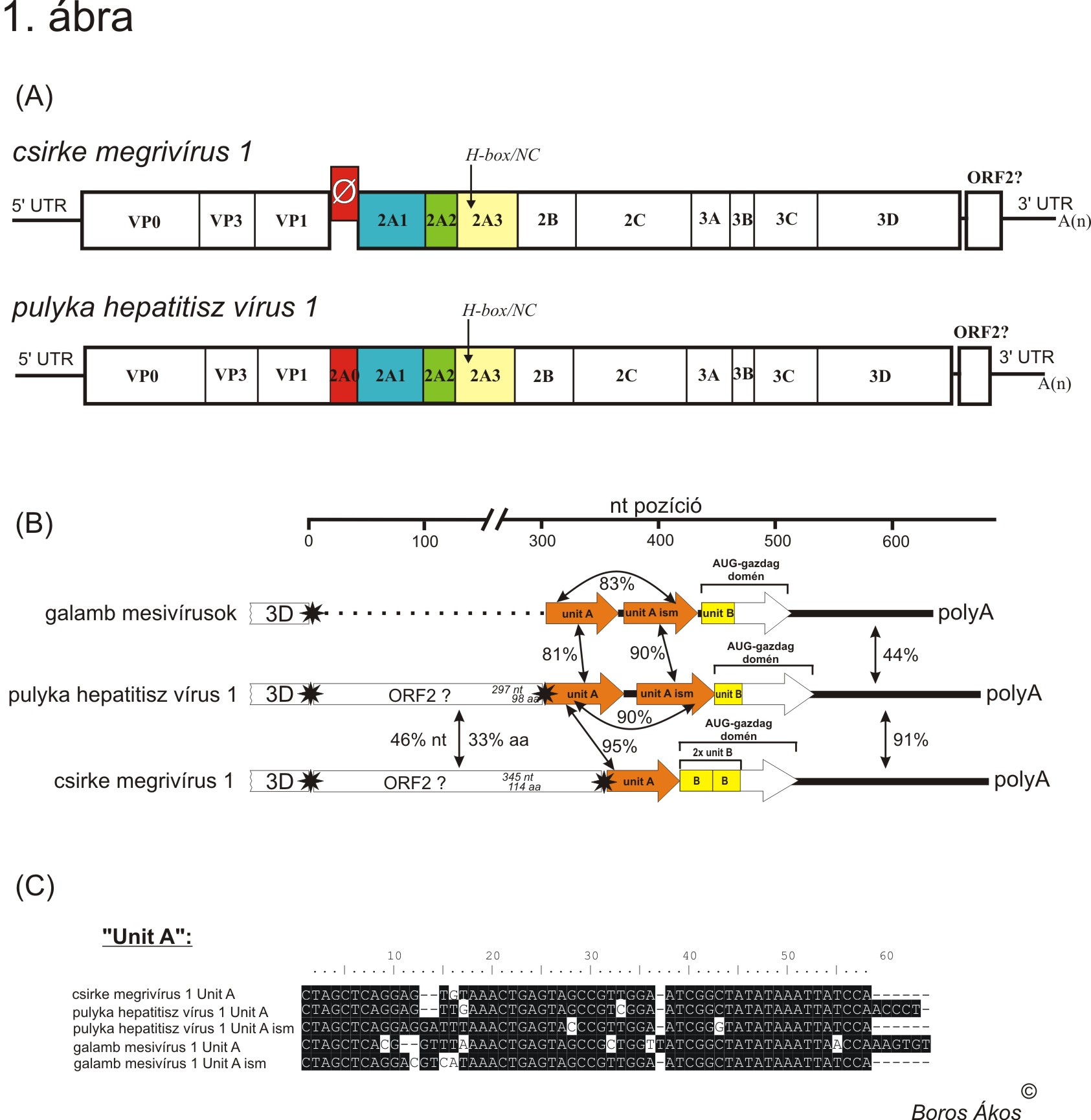
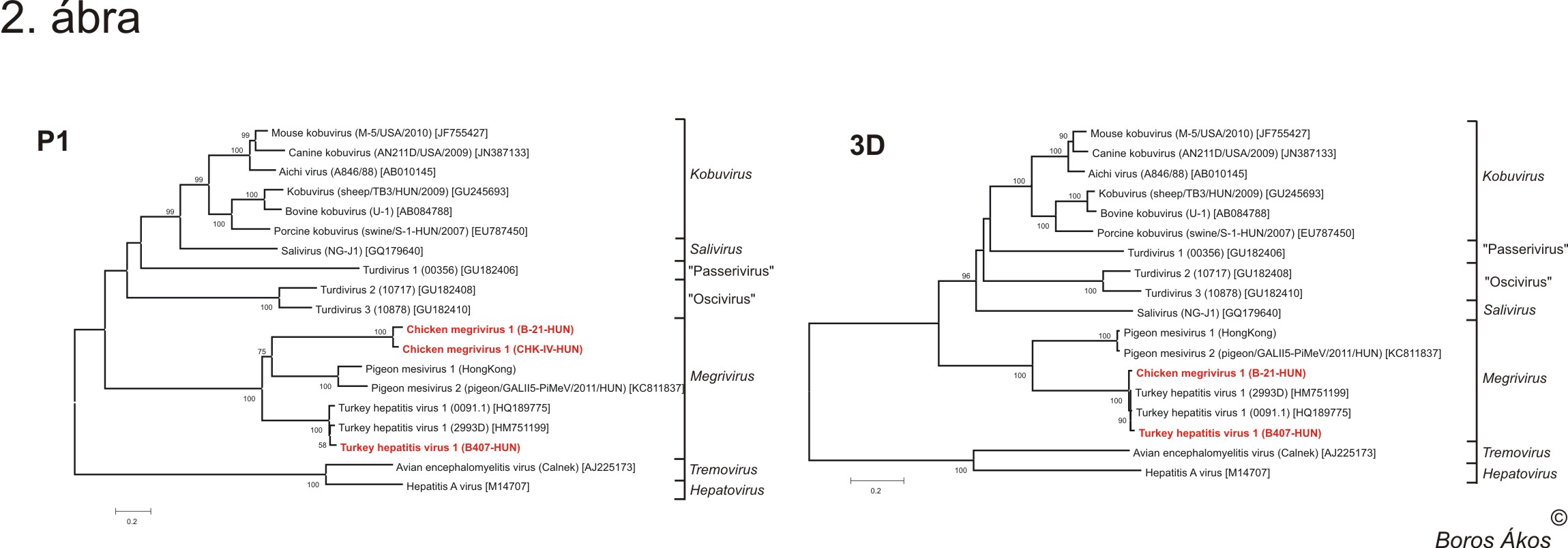
Ha megvizsgáljuk a THV1, a galamb mesivírusok és a csirke megrivírusok filogenetikai pozícióját a kapszid fehérjéket kódoló szakaszon (P1) és a konzervatív 3D RdRp (RNS-függő RNS polymeráz) kódoló szakaszon, akkor a csirke megrivírusokat különböző pozícioban találjuk a két fán (2. kép). A P1 régióban jól látható az elkülönülés a THV-1-től és a galamb mesivírusoktól, míg a 3D f fán a THV-1 és a csirke megrivírusok egy kompakt csoportot alkotnak (2. kép). A filogenetikai pozíciók különbsége a csirke megrivírusok (vagy a THV-1) evolúciós fejlődésében legalább egy, a 2A régióban bekövetkező rekombinációs eseményre utalnak. A feltételezett rekombinációs esemény két, nukleotid szekvenciában távoli (legalább külön fajba tartozó) vírus között jöhetett létre (Boros és mtsai., 2014). A fajok közötti rekombináció nyomai a jelenleg ismert picornavírusok között nagyon ritkák.
A negyedik megrivírus fajt nemrég azonosították kacsákból. A kacsa megrivírus RNS genomja a csirke megrivírusokhoz hasonlóan három különböző 2A fehérjét kódol. Érdekessége, hogy a szintén hosszú (>600 nukleotid) 3’ UTR-je nem mutat hasonlóságot a többi három megrívírussal, nem tartalmaz sem Unit A, sem Unit B szekvenciákat, sem AUG-gazdag régiót. Emellett a második ORF sem kimutatható. A kacsa megrivírus 3’ UTR szekvenciája inkább a kacsa hepatitisz A (Duck hepatitis A / DHAV) 3’ UTR-jével mutat rokonságot (Liao et al., 2014).
Referenciák
Boros, Á., Pankovics, P., Knowles, N.J., Nemes, C., Delwart, E., Reuter, G., 2014.Comparative complete genome analysis of chicken and turkey megriviruses (family Picornaviridae): long 3’ untranslated regions with a potential second open reading frame and evidence for possible recombination. J. Virol. 88, 6434-6443.
Farkas, T., Fey, B., Hargitt 3rd, E., Parcells, M., Ladman, B., Murgia, M., Saif, Y., 2012. Molecular detection of novel picornaviruses in chickens and turkeys. Virus Genes 44, 262-272.
Liao, Q., Zheng, L., Yuan, Y., Shi, J., Zhang, D., 2014. Genomic characterization of a novel picornavirus in Pekin ducks, Vet. Mic. http://dx.doi.org/10.1016/j.vetmic.2014.05.002
Phan, T.G., Vo, N.P., Boros, Á., Pankovics, P., Reuter, G., Li, O.T.W., Wang, C., Deng, X., Poon, L.L.M., Delwart, E., 2013. The viruses of wild pigeon droppings. PLoS ONE 8, e72787.
Wang, X., Liu, N., Wang, F., Ning, K., Li, Y., Zhang, D., 2014.Genetic characterization of a novel duck-origin picornavirus with six 2A proteins. J. Gen. Virol. 95, 1289-1296.
Honkavuori KS, Shivaprasad HL, Briese T, Street C, Hirschberg DL, Hutchison SK, Lipkin WI.2011. Novel picornavirus in turkey poults with hepatitis, California, USA. Emerg. Infect. Dis. 17:480–487.
Snoeyenbos GH, Bosch HI, Sevoian M. 1959. An infectious agent producing hepatitis in turkeys. Avian Diseases. 3:377-388.
A Picornaviridae család filogenetikus rendszere
2014.06.20. 10:42
A picornavírus család (Picornaviridae) jelenleg hivatalosan 46 fajból áll, amely összesen 26 különböző nemzetségbe tartozik. A hivatalos nemzetségek a következők: Aphthovirus, Aquamavirus, Avihepatovirus, Avisivirus, Cardiovirus, Cosavirus, Dicipivirus, Enterovirus, Erbovirus, Gallivirus, Hepatovirus, Hunnivirus, Kobuvirus, Megrivirus, Mischivirus, Mosavirus, Oscivirus, Parechovirus, Pasivirus, Passerivirus, Rosavirus, Salivirus, Sapelovirus, Senecavirus, Teschovirus és Tremovirus. A hivatalos faj és nemzetségszám folyamatosan, és egyre gyorsuló tempóban növekszik. Jelenleg legalább 18 lehetséges új picornavírus faj vár hivatalos besorolásra, amelyek legalább 7 további nemzetséggel fogják bővíteni a Picornaviridae családot. Az úgynevezett második generációs szekvenálás (amelyet szoktak még következő generációs szekvenálásnak - Next generation sequencing, vagy tömegszekvenálásnak - Mass sequencing is nevezni) megjelenésével és egyre szélesebb körű elterjedésével az új mikroorganizmusok, köztük az új (picorna)vírusok felfedezése is rohamtempóban gyorsul. A madarak picornavírusainak mind nagyobb számú leírása mellet alacsonyabb gerinces csoportok (hüllők, kétéltűek és halak) vírusainak genom-szintű vizsgálata is elkezdődött. A jelenleg ismert picornavírusok összesen öt különböző filogenetikai kládot alkotnak, amelyeket az alábbi képen (1. ábra) különböző színekkel és római számokkal jelöltem. Az ábrán dőlt betűvel vannak szedve a 26 hivatalos picornavírus nemzetségek nevei, amely írásmód egyben a hivatalosan elfogadott nemzetségek jelenleg érvényben lévő írásmódja is (1. ábra).
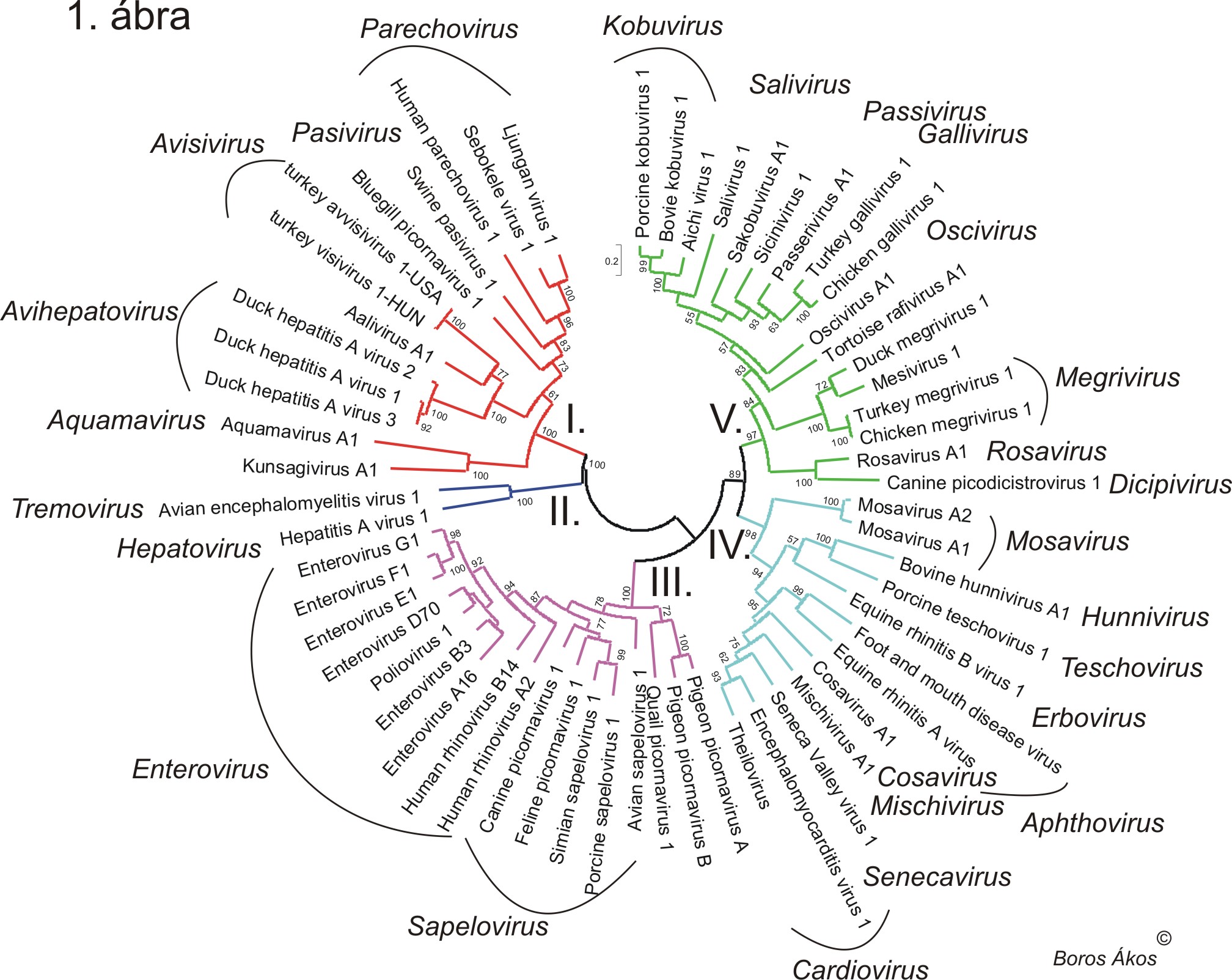
A különböző nemzetségekbe tartozó picornavírusokat többféle gerinces fajból izoláltak, azonban a fajok döntő többségét (> 80%-át) emlősökből azonosították. Az alábbi képen (2. ábra) a teljesség igénye nélkül próbáltam ábrázolni az egyes picornjavírusok gazdafajait (legalábbis azokat a fajokat, amelyekből az egyes vírusokat azonosították). Jól látható, hogy egy adott gazdafajnak számos különböző, törzsfejlődéstani szempontból távol eső picornavírusa is lehet. Jó példa erre az ember vagy a házi sertés különböző picornavírusainak filogenetikai elhelyezkedése: Emberi picornavírusok a család mind az öt ismert kládjában ismertek: I. klád: humán parechovírusok; II. klád: Hepatitisz A vírus; III. klád: Enterovírusok; IV. klád: Encephalomyocarditis vírus, Cosavírus; V. klád: Aichi vírus, Salivírus (2. ábra). Sertés picornavírusok is a II. klád kivételével mindenhol előfordulnak. Vannak olyan gerinces csoportok, amelyek látványosan alulreprezentáltak az alább látható gazdafaj-picornavírus törzsfán, ilyenek például a halak, vagy a hüllők, de például vannak olyan gerinces osztályok (!) pl. kétéltűek, amely csoportba hozzávetőlegesen 4800 állatfaj (!) tartozik, és amelynek jelenleg egyetlen ismert picornavírusa sincsen (2. ábra). Az alulreprezentáltság, illetve az egyes gerinces állatcsoportok picornavírusainak hiányának oka nem az, hogy az alacsonyabb rendű gerinceseknek nincsenek, vagy csak kevesebb picornavírusa van, sokkal inkább az, hogy ezeknek az állatcsoportoknak a vírusait csak mostanában kezdték el vizsgálni.
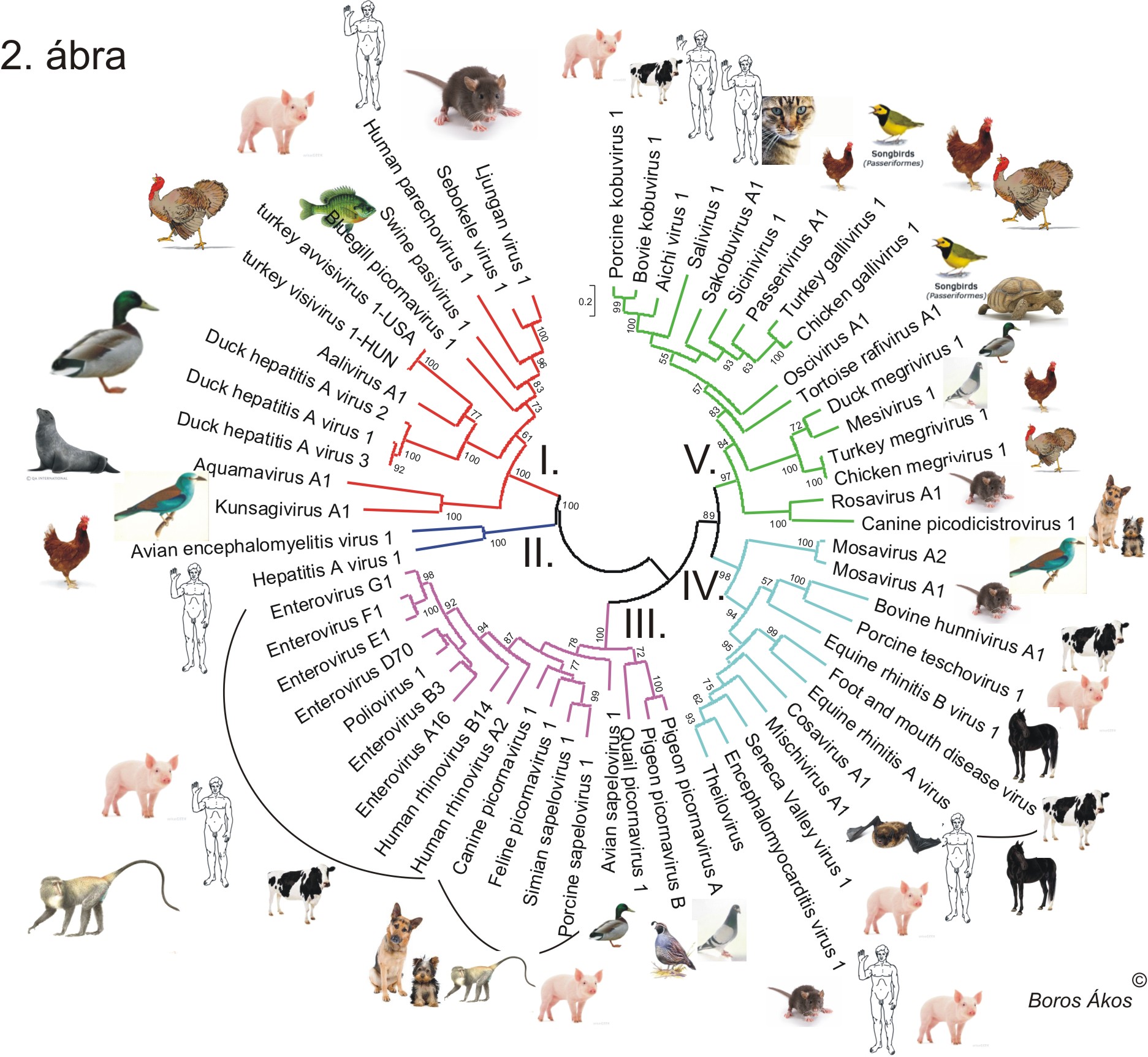
Szintén az ismert picornavírusok tekintetében az alulreprezentált gerincesek közé tartoznak a madarak is. 2006-ig összesen csak egy nem-emlős (madár) picornavírus, a házityúkokból (Gallus gallus domesticus) izolált, neurovirulens avian encephalomyelitis virus 1 (AEV-1) genomja volt ismert. 2010-re további két madár picornavírus faj teljes genomját határozták meg: a Duck hepatitis A virus (DHAV), amelyet korábban Duck Hepatitis virus 1 néven ismertek és az Avian sapelovirus 1 (ASV-1), amelyet leírásakor Duck picornavirus TW90A néven jelöltek. Mindkét vírust házi kacsákból (Anas platyrhynchos domestica) azonosítottak. Többek közt a fent említett tömegszekvenálási módszerek elterjedésének köszönhetően a madár picornavírusok száma az elmúlt pár évben rohamosan nő. Jelenleg összesen 19 madár picornavírus faj ismert, amelyek legalább 14 nemzetséget alkotnak (táblázat)
|
Nemzetség |
Faj |
Referencia törzs (Rövidítés) |
A referencia törzs GenBank azonosítószáma |
Irodalmi hivatkozás |
|
nem besorolt |
nem besorolt |
Quail picornavirus 1 (QPV-1) |
JN674502 |
Pankovics et al., 2012 |
|
nem besorolt |
nem besorolt |
Pigeon picornavirus B (PiPV-B) |
FR727144 |
Kofstad and Jonassen, 2011 |
|
Sapelovirus |
Avian sapelovirus |
Avian sapelovirus 1 (ASV-1) |
AY563023 |
Tseng and Tsai, 2007 |
|
Megrivirus |
Melegrivirus A |
turkey hepatitis virus 1 |
KF961188 |
Honkavouri et al.,2011; Boros et al., 2014; Farkas et al., 2012 |
|
nem besorolt |
chicken megrivirus 1 (ChMV-1) |
KF961186 |
Boros et al., 2014; Farkas et al., 2012 |
|
|
nem besorolt |
Mesivirus 1 (MeV-1) |
KC876003 |
Phan et al., 2013 |
|
|
Mesivirus 2 (MeV-2) |
KC811837 |
|||
|
nem besorolt |
duck megrivirus 1 (DuMV-1) |
KC663628 |
Liao et al., 2014 |
|
|
Gallivirus |
Gallivirus A |
gallivirus A1 (GV-A1) |
JQ691613 |
Boros et al., 2012; Farkas et al., 2012 |
|
nem besorolt |
chicken gallivirus 1 (ChGV-1) |
KF979337 |
Lau et al., 2014 |
|
|
"Sicinivirus" |
"Sicinivirus A" |
Sicinivirus 1 (SiV-1) |
KF741227 |
Bullman et al., 2014 |
|
Passerivirus |
Passerivirus A |
Passerivirus A1 (PasV-A1) |
GU182406 |
Woo et al., 2010 |
|
Oscivirus |
Oscivirus A |
Oscivirus A1 (OscV-A1) |
GU182408 |
|
|
Oscivirus A2 (OscV-A2) |
GU182410 |
|||
|
Avisivirus |
Avisivirus A |
Avisivirus A1 (AsV-A1) |
KC465954 |
Boros et al., 2013a |
|
KC614703 |
Ng et al., 2013 |
|||
|
nem besorolt |
chicken picornavirus 2 and 3 |
KF979333 |
Lau et al., 2014 |
|
|
Avihepatovirus |
Duck hepatitis A virus |
Duck hepatitis A virus 1 (DHAV-1) |
DQ249299 |
Tseng et al., 2007; Tseng and Tsai, 2007; Kim et al., 2007; |
|
Duck hepatitis A virus 2 (DHAV-2) |
EF067923 |
|||
|
Duck hepatitis A virus 3 (DHAV-3) |
DQ256132 |
|||
|
"Aalivirus" |
"Aalivirus A" |
Aalivirus A1 |
KJ000696 |
Wang et al., 2014 |
|
Tremovirus |
Avian encephalomyelitis virus |
Avian encephalomyelitis virus 1 (AEV-1) |
AJ225173 |
Marvil et al., 1999 |
|
"Kunsagivirus" |
nem besorolt |
"Kunsagivirus A1" (KuV-A1) |
KC935379 |
Boros et al., 2013b |
|
"Mosavirus" |
nem besorolt |
"Mosavirus A2" (MoV-A2) |
KF958461 |
Reuter et al., 2014 |
Az új madár picornavírusok megismeréséhez kutatócsoportunk is hozzájárult: az elmúlt két évben összesen 8 új madár picornavírus fajt azonosítottunk első ízben, amelyek négy új nemzetség (Gallivirus, Avisivirus, ”Kunsagivirus” és egy elnevezésre váró további nemzetség, amelyet a fürj picornavírus 1/Quail picornavirus 1 alkot) névadó törzsei lettek. A 8 új madár picornavírus fajt különböző vad és háziasított madárfajokból azonosítottuk: (i) fürjekből (Coturnix coturnix): fürj picornavírus 1/Quail picornavirus 1 (Pankovics és mtsai, 2012). (ii) szalakótákból (Coracias garrulus): Kunsagivirus A1 (Boros és mtsai, 2013a) és Mosavirus A2 (Reuter és mtsai., 2014). (iii) pulykákból (Meleagris gallopavo): Gallivirus A1/pulyka gallivírus 1 (Boros és mtsai., 2012) és Avisivirus A1/pulyka avisivirus 1 (Boros és mtsai., 2013b) és Turkey hepatitis virus 1/Turkey megrivirus 1/pulyka megrivírus 1 (Boros és mtsai., 2014). (iv) házigalambokból (Columba livia domestica): mesivírusok (Phan et al., 2013). (v) házi tyúkokból (Gallus gallus domesticus): Chicken megrivirus 1/csirke megrivírus 1 (Boros és mtsai., 2014). A fent felsorolt, a kutatócsoportunk által azonosított magyarországi madár picornavírusokat (Hungary végződésű nevek) az ismert madár picornavírusokkal együtt (piros nevek) az alábbi filogenetikai fán ábrázoltam (3. ábra).
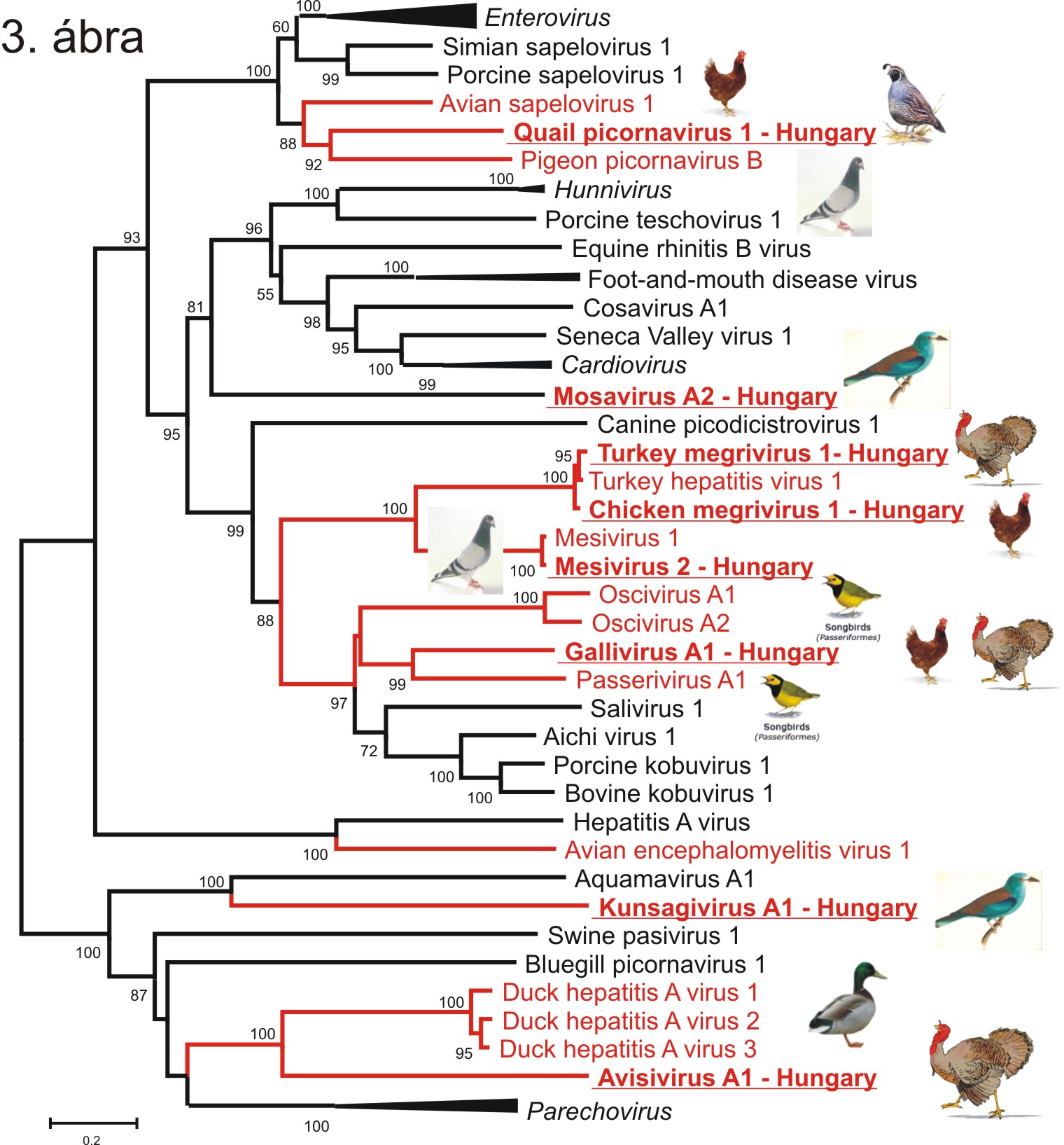
Az eddig azonosított madár picornavírusok filogenetikai elhelyezkedését tekintve igen változatos képet mutatnak, jelen vannak a picornavírusok mind az öt kládjában, azonban a IV. klád jelenleg egyetlen madár picornavírusa, a szalakótából azonosított ”Mosavírus A2” lehetséges, hogy a madár által elfogyasztott más állat (valamilyen kisemlős?) vírusa, és nem egy madárvírus.
Referenciák:
Boros, Á., Kiss, T., Kiss, O., Pankovics, P., Kapusinszky, B., Delwart, E., Reuter, G., 2013a. Genetic characterization of a novel picornavirus distantly related to the marine mammal-infecting aquamaviruses in a long-distance migrant bird species, European Roller (Coracias garrulus). J. Gen. Virol. 94, 2029-2035.
Boros, Á., Nemes, C., Pankovics, P., Kapusinszky, B., Delwart, E., Reuter, G., 2013b. Genetic characterization of a novel picornavirus in turkeys (Meleagris gallopavo) distinct from turkey galliviruses and megriviruses and distantly related to the members of the genus Avihepatovirus. J. Gen. Virol. 94, 1496–1509.
Boros, Á., Nemes, C., Pankovics, P., Kapusinszky, B., Delwart, E., Reuter, G., 2012.Identification and complete genome characterization of a novel picornavirus in turkey (Meleagris gallopavo). J. Gen. Virol. 93, 2171–2182.
Boros, Á., Pankovics, P., Knowles, N.J., Nemes, C., Delwart, E., Reuter, G., 2014.Comparative complete genome analysis of chicken and turkey megriviruses (family Picornaviridae): long 3’ untranslated regions with a potential second open reading frame and evidence for possible recombination. J. Virol. 88, 6434-6443.
Bullman, S., Kearney, K., O' Mahony, M., Kelly, L., Whyte, P., Fanning, S., Morgan, J.G., 2014. Identification and genetic characterisation of a novel picornavirus from chickens. J. Gen. Virol. In press. doi: 10.1099/vir.0.061085-0.
Farkas, T., Fey, B., Hargitt 3rd, E., Parcells, M., Ladman, B., Murgia, M., Saif, Y., 2012. Molecular detection of novel picornaviruses in chickens and turkeys. Virus Genes 44, 262-272.
Kim, M.C., Kwon, Y.K., Joh, S.J., Kim, S.J., Tolf, C., Kim, J.H., Sung, H.W., Lindberg, A.M., Kwon, J.H., 2007.Recent Korean isolates of duck hepatitis virus reveal the presence of a new geno- and serotype when compared to duck hepatitis virus type 1 type strains. Arch. Virol. 152, 2059-2072
Kofstad, T., Jonassen, C.M., 2011. Screening of feral and wood pigeons for viruses harbouring a conserved mobile viral element: characterization of novel astroviruses and picornaviruses. PLoS One 6(10):e25964.
Liao, Q., Zheng, L., Yuan, Y., Shi, J., Zhang, D., 2014. Genomic characterization of a novel picornavirus in Pekin ducks, Vet. Mic. http://dx.doi.org/10.1016/j.vetmic.2014.05.002
Marvil, P., Knowles, N.J., Mockett, A.P.A., Britton, P., Brown, T.D.K., Cavanagh, D., 1999. Avian encephalomyelitis virus is a picornavirus and is most closely related to hepatitis A virus. J. Gen. Virol. 80, 653-662.
Ng. T.F., Cheung, A.K., Wong, W., Lager, K.M., Kondov, N.O., Cha, Y., Murphy, D.A., Pogranichniy, R.M., Delwart, E., 2013. Divergent picornavirus from a Turkey with gastrointestinal disease. Genome Announc. 2, e00134-13.
Pankovics, P., Boros, Á. and Reuter, G., 2012. Novel picornavirus in domesticated common quail (Coturnix coturnix) in Hungary. Arch. Virol. 157, 525-530.
Phan, T.G., Vo, N.P., Boros, Á., Pankovics, P., Reuter, G., Li, O.T.W., Wang, C., Deng, X., Poon, L.L.M., Delwart, E., 2013. The viruses of wild pigeon droppings. PLoS ONE 8, e72787.
Reuter, G., Boros, Á., Kiss, T., Delwart, E., Pankovics, P., 2014. Complete genome characterization of mosavirus (family Picornaviridae) identified in dropping of European roller (Coracias garrulus) in Hungary. Arch. Virol, DOI 10.1007/s00705-014-2113-4
Tseng, C.H., Tsai, H.J., 2007. Sequence analysis of a duck picornavirus isolate indicates that it together with porcine enterovirus type 8 and simian picornavirus type 2 should be assigned to a new picornavirus genus. Virus Res.129, 104-114
Tseng, C.H., Knowles, N.J., Tsai, H.J., 2007. Molecular analysis of type 1 duck hepatitis virus indicated that it should be assigned to a new genus. Virus Res. 123, 190-203.
Wang, X., Liu, N., Wang, F., Ning, K., Li, Y., Zhang, D., 2014.Genetic characterization of a novel duck-origin picornavirus with six 2A proteins. J. Gen. Virol. 95, 1289-1296.
Woo, P.C., Lau, S.K., Huang, Y., Lam, C.S., Poon, R.W., Tsoi, H.W., Lee, P., Tse, H., Chan, A.S., Luk, G., Chan, K.H., Yuen, K.Y. 2010. Comparative analysis of six genome sequences of three novel picornaviruses, turdiviruses 1, 2 and 3, in dead wild birds and proposal of two novel genera, Orthoturdivirus and Paraturdivirus, in Picornaviridae. J. Gen. Virol. 91, 2433-2448.
A picornavírusok 5’ nem kódoló régióinak jellemzése
2014.05.12. 10:14
A picornavírusok (és egyéb más pozitív egyszálú RNS vírusok, pl. Hepatitisz C - Flaviviridae család) 5’ nem kódoló régiója (5’ UTR = UnTranslated Region) komplex másodlagos struktúrával rendelkezik, amely funkcionális szempontból két különböző régióra osztható. Az 5’ vég olyan strukturális elemeket tartalmaz, amelyek nélkülözhetetlenek a virális genom megsokszorozódásához. A transzláció iniciációs kodon (start kodon) előtti szakasz az ún. belső riboszóma kötőhely (IRES=Internal Ribosomal Entry Site) a virális poliprotein képződésének megindításáért felelős régió, amely a riboszómális komplex vírusgenomhoz történő kapcsolódását biztosítja. Régóta ismert, hogy az eukarióta mRNS molekulák transzlációja úgynevezett CAP-függő folyamat. (CAP azaz ”sapka” az érett eukarióta mRNS molekulák 5’ végéhez kovalensen kötött 7-metil-guanozin trifoszfát molekula, amely egyrészt védi az mRNS-t az RNS bontó enzimektől, másrészt transzláció iniciációs faktor-kötőhelyként szolgál. Bővebb információ az alábbi linken). Az eukarióta mRNS molekulákkal ellentétben a picornavírusok (és egyes egyéb más pozitív egyszálú RNS vírusok, mint például a Hepatitisz C) virális poliprotein kódoló régiójának transzlációja CAP-független, mivel a picornavírus genom 5’ végéhez nem kapcsolódik CAP, csak egy VPg peptid, amelynek funkciója az RNS védelmét kivéve teljesen eltér a CAP-tól. Továbbá egyes picornavírusok, mint például az enterovírusok a gazdasejtbe bejutva éppen a CAP-függő transzlációhoz szükséges iniciációs faktorokat (pl. eIF-4G) bontanak el, leállítva ezáltal a gazdasejt saját CAP-függő fehérje-gyártó apparátusát. Bővebben lásd a 2014.03.28.-ai bejegyzésemet. A picornavírus 280 és 450 nukleotid hosszúságú IRES régiója komplex másodlagos RNS-struktúrával rendelkezik és számos a gazdasejt által termelt iniciációs faktor (IF) és riboszómális komplex kötőhelyéül szolgál. Az IRES-IF-riboszóma komplex működése, ezáltal a virális poliprotein képződése teljesen CAP-független. Az IRES régió másodlagos RNS struktúrája alapján összesen 5 különböző picornavírus IRES típust (IRES I-V) azonosítottak: Az enterovírus-típusú IRES-I; a parecho-, cardio-, erbo- és aphthovírus típusú IRES-II; a hepatitis A-típusú IRES-III; a Hepacivírus/Pestivírus-típusú IRES-IV valamint a 2012-ben leírt kobu- és salivírus-típusú IRES-V. A felsorolásban szereplő víruscsoportokról, a picornavírusok taxonómiájáról bővebben olvashattok egy későbbi bejegyzésemben. A több doménes (összetett, kompakt struktúra), számos hajtű- és hurok elemből felépülő IRES típusok első ránézésre is eltérő másodlagos RNS-struktúrával rendelkeznek, lásd az alábbi ábrán. A doméneket általában római számokkal, az azokat alkotó hajtű-struktúrákat pedig betűkkel jelölik. A több doménes IRES legfontosabb funkciójú, és struktúrálisan legkonzervatívabb úgynevezett mag-doménjei (a 40S riboszómális alegység kötőhelye) közvetlenül a start kodon (AUG) előtt találhatóak.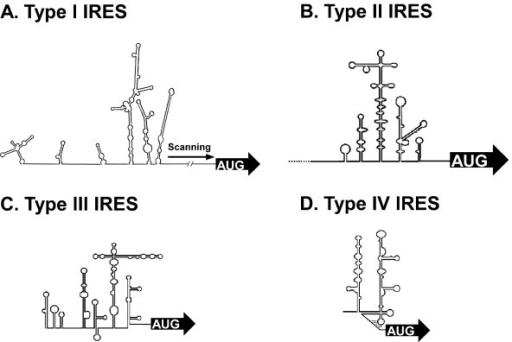
1. ábra: A különböző IRES típusok (I-IV) strukturális jellemzői. AUG = Transzláció iniciációs kodon.
Az eddig ismert emlős picornavírus fajok között a leggyakoribb az ún. IV-es típusú IRES. A 2. ábrán is jól látható, hogy a IV-es IRES mag-doménjeinek mérete, az azokat alkotó bázisok száma között lehetnek kisebb-nagyobb eltérések, de a domének fő szerkezeti vonásai konzervatívak.
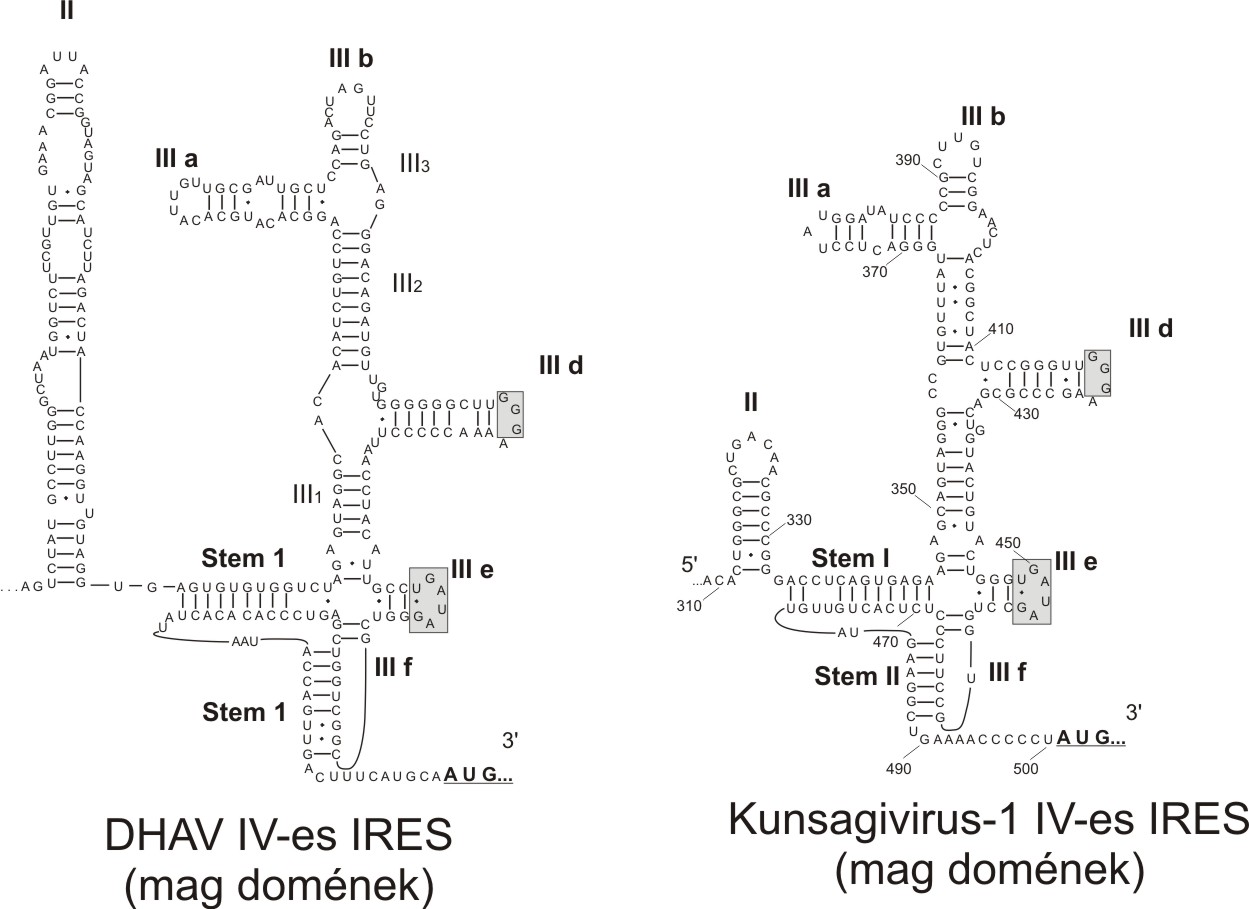
2. ábra: A IV-es típusú IRES mag doménjeinek másodlagos RNS-szerkezete. Az RNS bázisok közötti egyszerű vonalak a kanonikus bázispárokat, a fekete pontok az úgynevezett lötyögős (”wobble”) bázispárokat jelölik. Az egyes domének különböző méretei (pl. a domén II) ellenére a másodlagos szerkezet alapjában véve ugyanolyan a két IRES esetében.
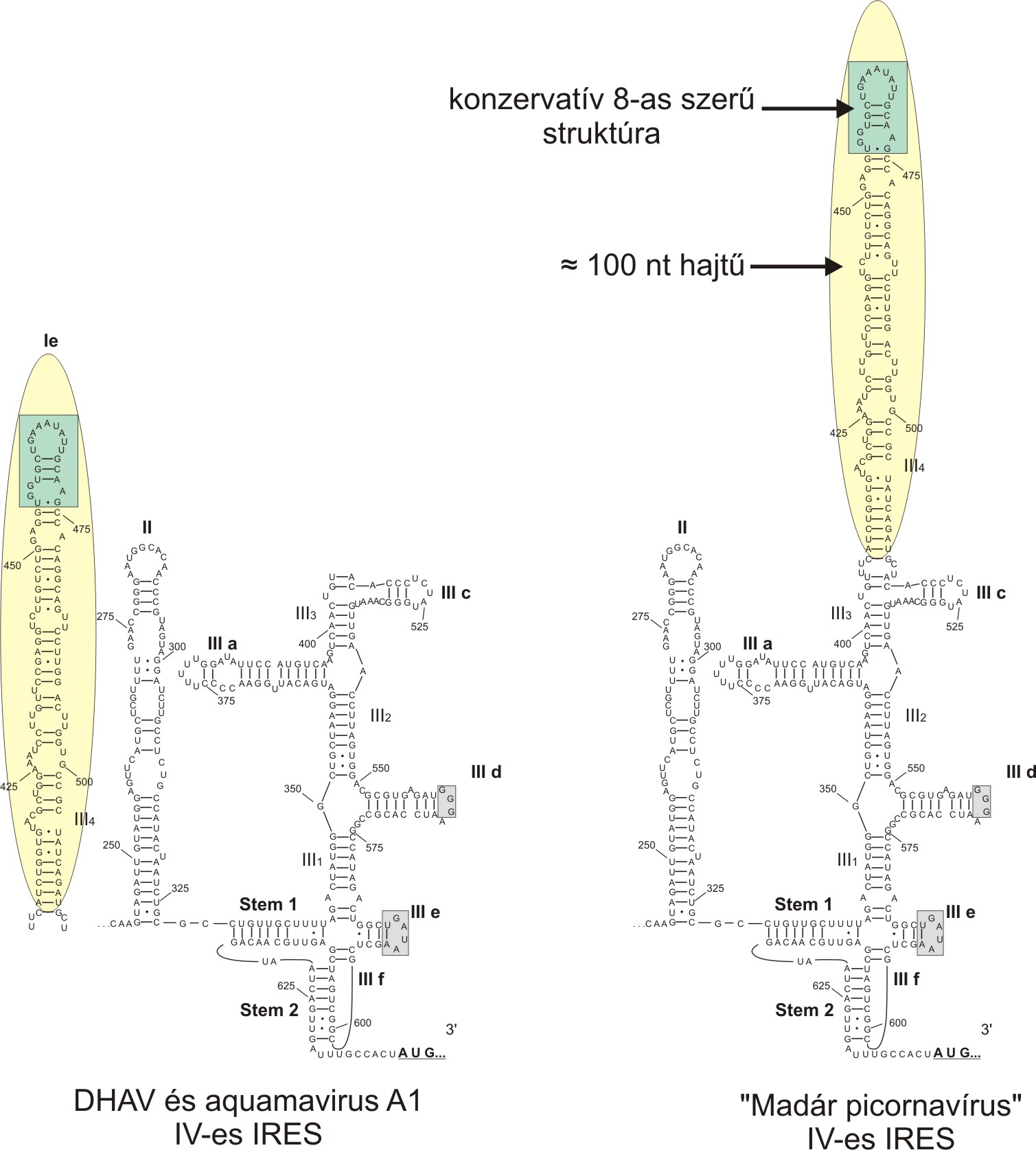
Az emlős picornavírusokhoz hasonlóan az eddig azonosított madár picornavírusok között is a IV-es típusú IRES előfordulása a leggyakoribb. Azonban az emlős IV-es IRES-el ellentétben a legtöbb madár picornavírus IV-es IRES tartalmaz egy kb. 100 nukleotidból álló hajtű-struktúrát, amelynek a csúcsi (apikális) részén egy 20 nukleotidból álló, szekvencia és másodlagos struktúra szintjén is konzervatív, 8-as számot formázó struktúra található (3. ábra). Ez a kb. 100 nukleotidos hajtű általában IV-es IRES III-as doménjének csúcsi részén található, azonban a kacsa hepatitisz A vírusoknál (duck hepatitis A virus = DHAV; Avihepatovirus nemzetség) ez a hosszú hajtű nem a III-as doménen, hanem külön doménként a II-es domén előtt (Ie) található (3. ábra). Eddigi ismereteink szerint a 100 nukleotidos hajtű a 8-as szerű csúcsi résszel a fókákat fertőzi képes Aquamavírus A1 (Aquamavirus nemzetség) kivételével csak a madár picornavírusok jellemzője, és csak a IV-es IRES részeként fordul elő. Az Aquamavírus A1 esetében a DHAV-hoz hasonlóan a 100 nukleotidos hajtű a II. domén előtt található. További érdekesség, hogy nem minden madár picornavírus IV-es típusú IRES-ének képezi részét a fent említett 100 nukleotidos hajtű. Például hiányzik az madár enkefalomielitisz vírus (avian encephalomyelitis virus = AEV; Tremovirus nemzetség); a madár sapelovírus (ASV-1; Sapelovirus nemzetség) és a nemrég leírt Kunsagivírus 1 (”Kunsagivirus” nemzetség) IV-es IRES-eiből. A pontos funkciója a 100 nukleotidos hajtűnek jelenleg ismeretlen, habár a DHAV IRES-en végzett deléciós vizsgálatok kimutatták, hogy az Ie domén (ami megfelel a 100 nukleotidos hajtűnek a 8-as szerű csúcsi résszel) struktúrájának megváltoztatása 40%-al csökkentette az IRES aktivitását. További részletekért lásd Pan és mtsai, 2011: J. Virol. 2012, 86(2):1129. DOI: 10.1128/JVI.00306-11. Mindenesetre a 100 nukleotidos hajtű-mentes IV-es IRES-ek jelenléte az emlős és egyes madár picornavírusok között a hosszú hajtű fontos, de nem-esszenciális funkciójára utalhatnak.
3. ábra: IV-es IRES madár picornavírus változatának másodlagos struktúrája. A 100 nukleotidos (nt) hajtű (sárga háttér) a konzervatív a 8-as szerű csúcsi résszel (zöld háttér) két különböző pozícióban helyezkedhet el.
Referenciák:
Pankovics, P., Boros, A. and Reuter, G., 2012. Novel picornavirus in domesticated common quail (Coturnix coturnix) in Hungary. Arch. Virol. 157, 525-530.
Boros, Á., Kiss, T., Kiss, O., Pankovics, P., Kapusinszky, B., Delwart, E., Reuter, G., 2013. Genetic characterization of a novel picornavirus distantly related to the marine mammal-infecting aquamaviruses in a long-distance migrant bird species, European Roller (Coracias garrulus). J. Gen. Virol. 94, 2029-2035.
Boros, Á., Pankovics, P., Knowles, N.J., Nemes, C., Delwart, E., Reuter, G., 2014. Comparative complete genome analysis of chicken and turkey megriviruses (family Picornaviridae): long 3’ untranslated regions with a potential second open reading frame and evidence for possible recombination. J. Virol. 88(11):6434.DOI: 10.1128/JVI.03807-13.
Összefoglaló a 18. Nemzetközi Picornavírus találkozóról
2014.04.14. 15:02
A kétévente megrendezésre kerülő picornavírus vándor konferenciát idén Belgium egyik csendes az Északi-tenger partján fekvő kis városában, Blankenbergben, a Floreal Hotel Konferencia Központban rendezték meg március 9 és 14. között. A konferencia hivatalos nyelve az angol volt. Közel 300 résztvevőjével a picornavírus találkozó a kisebb, ezáltal családiasabb légkörű konferenciák közé tartozik. A rendezvénysorozat hivatalos célja hogy a picornavírusokkal foglalkozó kutatók, orvosok és állatorvosok bemutathassák legfrissebb tudományos eredményeiket; megbeszélhessék, kicserélhessék tapasztalataikat, és hogy új, nemzetközi együttműködéseket köthessenek. A viszonylag alacsony létszám ellenére a világ számos országából, többek közt Angliából, Egyesült Államokból, Etiópiából, Kínából, Japánból és Ausztráliából is érkeztek résztvevők. Előadóként egyedül én képviseltem hazánkat. A konferencián lehetőségem volt több, eddig csak a tudományos közleményeikből ismert szakemberrel személyesen is találkozni. A konferencia lineáris felépítését tekintve eltérő a legtöbb hazai és nemzetközi konferenciától, ahol az előadásokat és a tudományos posztereket párhozamosan futó szekciókra bontják, ezáltal az előadások rangsorolására, fontosnak tartott, egy időben zajló előadások kihagyására kényszerítve a résztvevőket. A hat napos konferencia programjának lineáris felépítése lehetővé tette a résztvevők számára, hogy a konferencia minden előadását meghallgassák. A konferencián három különböző előadás-típussal lehetett találkozni: a ”Vitaindító” előadással (Keynote lecture), amelyek a szekciók fő témáit bemutató, 30 perces előadások voltak; szekciós előadásokkal (Communications), melyek az egyes szekciók tudományos érdeklődésre számot tartó 8 illetve 12 perces előadásai voltak; illetve egy-egy szűkebb témát tömören bemutató, rövid 5 perces (Shotgun presentation) előadásokkal. Az 5 perces előadások anyagát kötelezően a poszter-szekcióban is be kellett mutatni. Az a megtiszteltetés ért, hogy a konferenciára benyújtott anyagomat egy 8 perces szekciós-előadás keretében mutathattam be. Az előadásom címe: Avian picornaviruses: detection, characterization and comparative genome analysis, amelyet kedden (03.11) a 3. ”Virus genetics, genome plasticity, novel viruses, classification” témájú szekcióban adhattam elő. A találkozó tematikáját, amelynek fókuszában a találkozó nevéből is adódóan a picornavírusok (Picornaviridae víruscsalád) álltak, az egyes szekciók témái jól jellemzik. Az előadások és tudományos poszterek az alábbi főbb témákkal foglalkoztak: (i) Vírusok életciklusa, replikációja, transzlációja; (ii) Vírus-gazdasejt kölcsönhatás; (iii) Virális genomika, plaszticitás, új vírusok, nevezéktan; (iv) Epidemiológia, klinikai virológia és új diagnosztikai módszerek; (v) Antivirális szerek, vakcinák, betegség-megelőzés és kontroll, eradikáció. Az előadások és a poszterek megtekintése után több fontos észrevételt is tettem. A találkozón bemutatott munkák döntő többsége többszereplős, nemzetközi együttműködések eredményein alapuló, főleg humán-egészségügyi vagy állategészségügyi/gazdasági szempontból fontos picornavírusok vizsgálatáról szóltak. Jelentős erőfeszítéseket tesznek egyes emberi kórokozók, úgy mint az akut hepatitiszt okozó hepatitisz A vírus, többek közt meningoencephalitis-t is okozó Enterovírus 71, a nemrég felfedezett Rhinovírus C vírusok, illetve a páros ujjú patások között ragadós száj- és körömfájást okozó vírus (foot-and-mouth disease virus, FMDV) virális életciklusának, patogenitásának részletekbe menő feltárására. Emellett több, a humán parechovírusok, illetve a különböző enterovírusok epidemiológiai vizsgálatával, molekuláris diagnosztikájával, esetleges antivirális kezelési módszereivel foglalkozó posztert is találtam. Laboratóriumi vizsgálatok ígéretes eredményeket mutattak az endoplazmatikus retikulumban és a Golgi komplexben előforduló foszfatidilinozitol 4-kináz III béta enzim-blokkolók, illetve virion kicsomagolódását gátló, antivirális hatású imidazolidin-vegyületek, illetve a vapendavir enterovírus fertőzésekben történő alkalmazhatóságáról. Ezen antivirális szerek vizsgálata még csak laboratóriumi stádiumban van. Érdekességként jelzem, hogy a régóta ismert humán kórokozók (lásd feljebb) vizsgálata mellett új potenciálisan humán patogén vírusok, például a nemrég azonosított (2007) első humán kardiovírus, a Saffold vírus (Cardiovirus nemzetség) patogenitásának vizsgálatai is megkezdődtek.
Az egyik legérdekesebb előadást dr. Mark Pallansch, a CDC (Centers for Disease Control and Prevention Atlanta, Georgia, USA) Virológiai részlegének igazgatója adta elő a globális poliovírus eradikáció jelenlegi állásáról, amelynek címe: ”The Long and Winding Road to Polio Eradication: How close we are?”. Az 1988-ban létre hozott, a járványos gyermekbénulást okozó poliovírus világ szinten történő kiirtását célzó program 2000-re becsülte a poliovírus eradikációjának végdátumát. Sajnos még 2013-ban is világ szinten mintegy 400 jelentett polio esetről tudunk. A teljes eradikáció még évekre van, azonban a program eddig is jelentős sikereket ért el: a kettes-típusú vad poliovírus kiirtásra került, és 3-as típusú vad poliovírus okozta megbetegedést sem jelentettek az elmúlt 3 évben, azonban több vakcina-eredetű 2-es poliovírus okozta járvány is bejelentésre került az elmúlt években. Nagy sikerként könyvelhető el egyrészt, hogy Indiában 3 éve nem volt vad poliovírus okozta bejelentett megbetegedés, másrészt hogy a teljes délkelet-ázsiai régiót polio-mentesnek nyilvánították. A program számára a legnagyobb kihívást az összesen háromra szűkült vad poliovírus endémiás országok (Afganisztán, Pakisztán és Nigéria) polio-mentesítése, illetve a vad poliovírusoknak az eddig polio-mentes területeken (pl. Szomália, Szíria illetve Izrael) részben az endémiás területekről történő újra terjedése jelenti. Mivel az orális vakcina (OPV) törzsek okozta polio-járványok aránya egyre számottevőbb, ezért az OPV vakcinák fokozatos kivezetése és az inaktivált polio vakcinák (IPV) bevezetése, valamint a monitoring rendszerek (környezeti minták, szennyvíz, illetve élő vizek poliovírus vizsgálata) felállítása/fenntartása és a vakcináció folytatása az endémiás országokban a program jelenlegi legfőbb feladatai.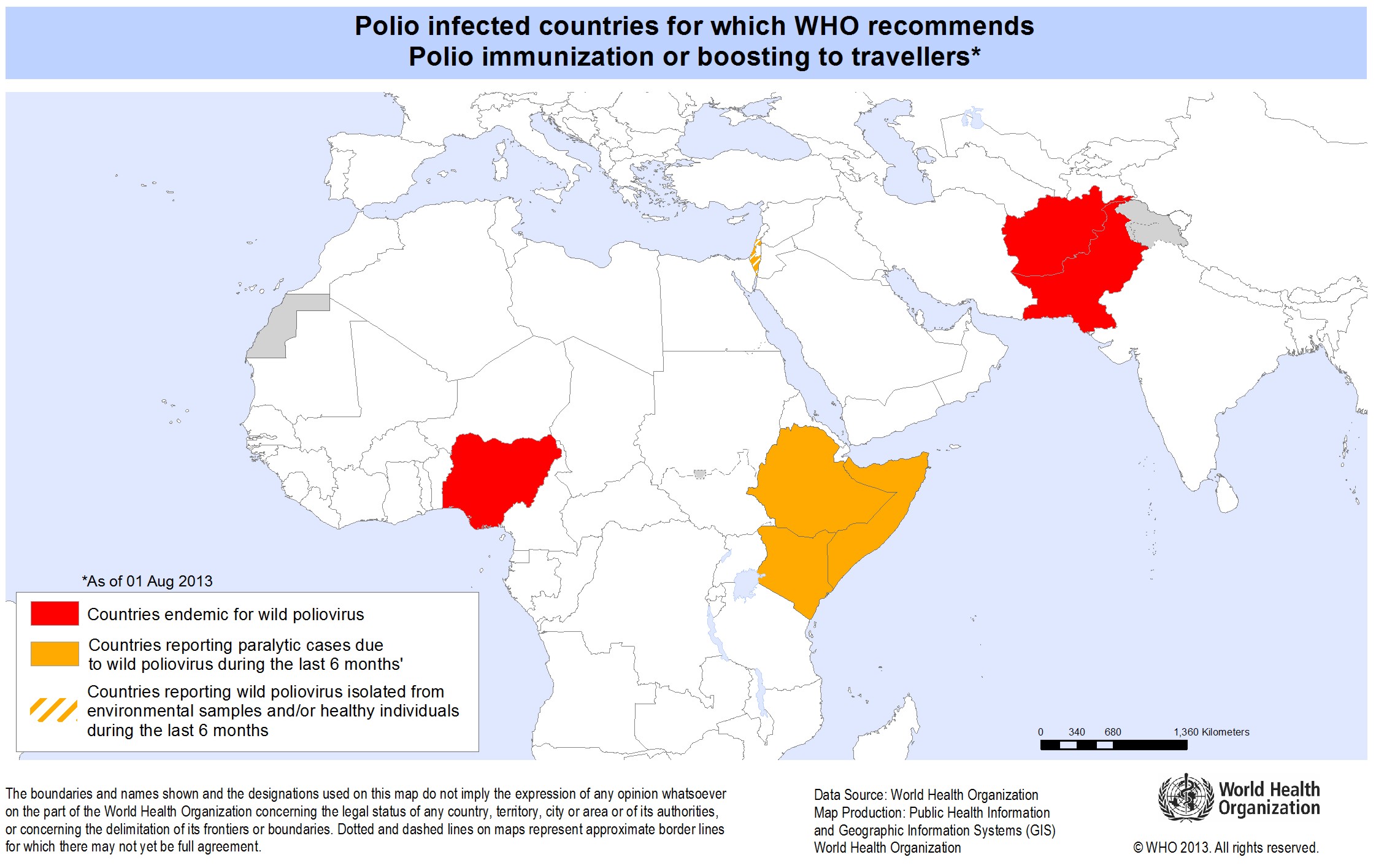
A poliovírus (remélhetően mielőbbi) eradikációja után több kutató szerint az úgynevezett neurovirulens Enterovírus 71 (EV-71) lehet az egyik legfontosabb népegészségügyi probléma kiváltképp a távol-keleti országokban, hívta fel a figyelmet rá Satoshi Koike az ”Enterovirus 71: an emerging enterovirus” című előadásában. Főleg a csecsemők és kisgyermekek körében úgynevezett kéz-láb-száj betegséget okozó EV-71 az esetek egy részében olykor súlyos neurológiai tünetekkel (neurovirulens EV-71) is járhat. A neurovirulens EV-71 főleg Délkelet- és Kelet-Ázsia országaiban (Tajvan, Malajzia, Szingapúr, Japán és Kína) okoznak jelentős közegészségügyi problémát. Napjaink egyik fontos felfedezése a neurovirulens EV-71-el kapcsolatban a Scavenger receptor B2, mint vírus-receptor szerepe a neurológiai tünetekkel járó EV-71 fertőzésekben.
A konferencia másik érdekes előadása Prof. Stan Lemon-nak a hepatitisz A vírus immunrendszer kikerülését célzó, eddig nem ismert stratégiájának bemutatásáról szólt (”Opening lecture: Hepatitis A virus”). A közel egy órás előadás lényegében Lemon és munkatársainak a tavalyi évben a Nature című tudományos folyóiratban megjelent (Nature 2013 April 18; 496(7445): 367–371.) publikációjára épült. Az úgynevezett lipid burok nélküli hepatitisz A vírussal (Picornaviridae család, Hepatovirus nemzetség) fertőződött májsejtekből a virionok képesek a gazdasejt membránjával körülvett, exoszómához hasonló vezikulákba csomagolódva az intracelluláris térbe, illetve a véráramba kiürülni. 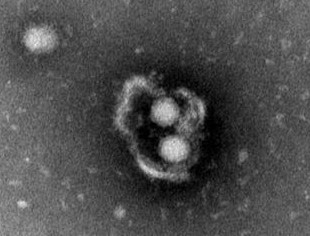
A membránnal határolt vezikulák egyszerre több (2-3 db.) fertőzőképes viriont is tartalmazhatnak, amelyekhez nem képesek kapcsolódni a gazdaszervezet neutralizáló antitestjei, ezáltal (részlegesen) kibújva az immunrendszer blokkoló hatása alól. Az úgynevezett burkos hepatitisz A vírus (eHAV = enveloped HAV) képződésének hátterében a HAV 2A fehérjéje áll, amely a többi picornavírussal ellentétben nem enzimatikus, hanem strukturális funkcióval rendelkezik.
A picornavírus találkozó lehetőséget biztosít többek közt a picornavírusok jelenlegi és jövőbeni rendszertanának megvitatására, új nemzetségek, fajok nevezéktan-javaslatainak megtárgyalására. A találkozón Nick. J. Knowles az ICTV (International Committee on Taxonomy of Viruses) Picornavirus Study Group jelenlegi vezetője mutatta be a picornavírus család jelenlegi rendszertanát és az újonnan felfedezett vírusok rendszertani besorolásának, az új rendszertani egységek elnevezéseinek javaslatát a ”Picornaviridae: 26 genera, 46 species and growing” című előadásában. Jelenleg érvényes hivatalos rendszertani besorolások alapján a Picornaviridae víruscsalád 46 fajból áll, amelyet összesen 26 különböző nemzetségbe soroltak. A hivatalosan elfogadott picornavírus fajokon kívül legalább 18 új, lehetséges picornavírus fajról tudunk, amelyek legalább 7 új nemzetséget alkotnak. Ezeket az elmúlt pár évben azonosították, és hivatalos rendszertani besorolásuk még nem történt meg. A picornavírusok rendszertana egyre komplexebb, az új nemzetségek egyre rohamosabban növekvő száma (a nemzetségek száma kétévente szinte megduplázódik) miatt új, nemzetségek fölötti rendszertani egységek, úgynevezett alcsaládok létrehozására tettek javaslatot. Az esetleges alcsaládok száma, nevezéktana és nemzetség-összetétele még kidolgozás alatt áll. Emellett az eddig hivatalosan érvényben nemzetségek jövőbeli ”újrafogalmazása” is felmerült.
A következő picornavírus találkozót 2016. szeptember 4 és 8 között Svájcban rendezik meg.