Rendhagyó madár picornavírusok bemutatása 3. rész
2014.09.22. 15:24
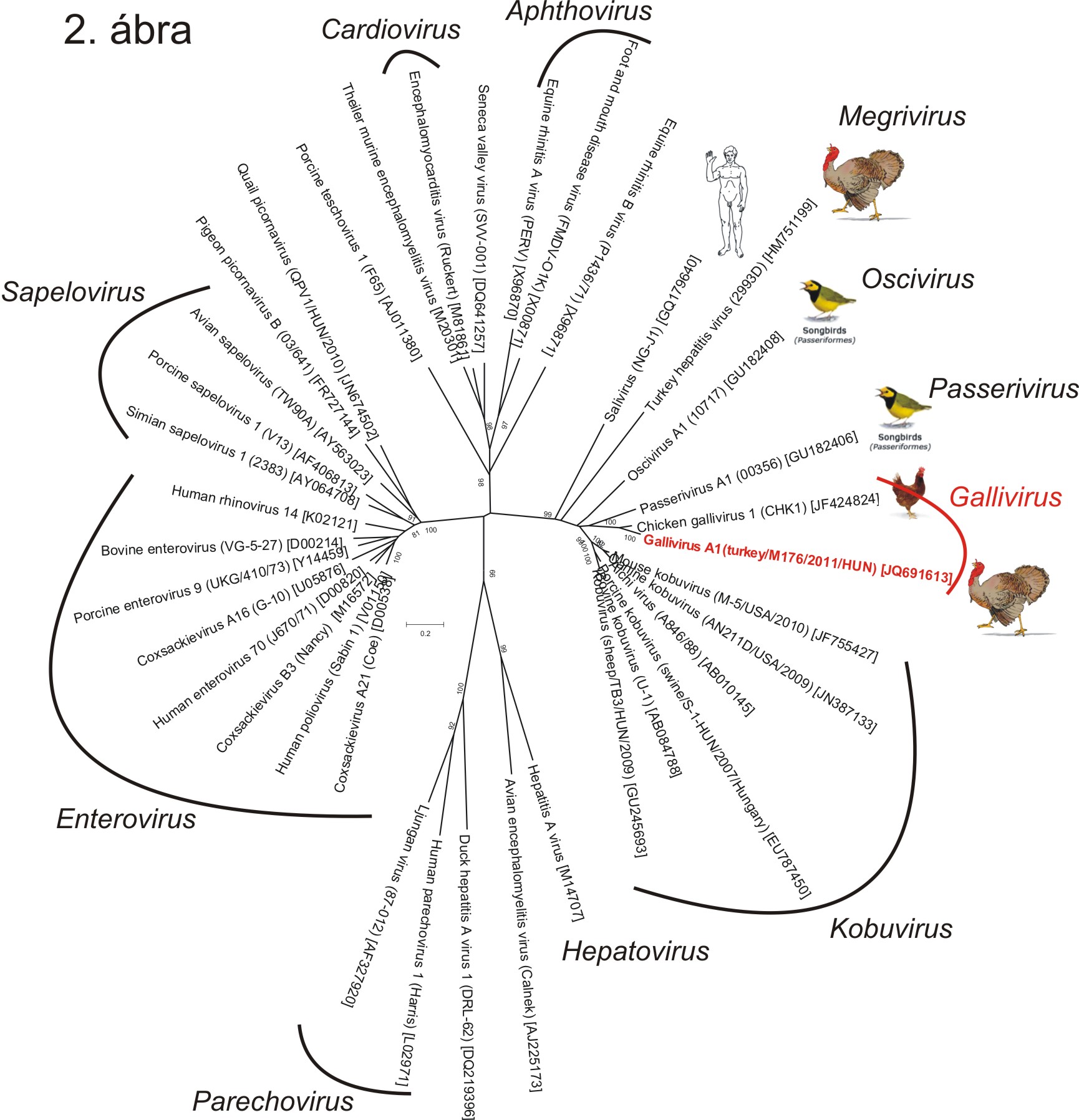
Farkas és munkatársai 2012-ben több, az énekesmadarakból azonosított turdivírusokkal (jelenleg passeri-, és oscivírusok), illetve az emlősökből azonosított kobuvírusokkal (Kobuvirus nemzetség) távoli rokon picornavírust azonosítottak pulyka és csirke székletmintákból, amelyeket gallivírusként neveztek el (Farkas és mtsai., 2012). Konvencionális RT-PCR módszereket alkalmazva csak részleges, a vírusgenom P3 régiójából származó szekvenciákat határoztak meg és tettek elérhetővé a GenBankban (JF424824-30). Kutatócsoportunknak magyarországi pulykamintákból virális metagenomikai és RT-PCR módszerekkel, még szintén 2012-ben sikerült a pulyka gallivírus teljes RNS genomjának meghatározása (Boros és mtsai., 2012). A pulyka gallivírus genomja 8496 nukleotid (nt) hosszú a 3’ végén lévő poliA-farok nélkül. A genom egy 7425 nt (2474 aminosav/aa) hosszú nyílt leolvasási keretet (ORF) tartalmaz. A vírusgenom egy N-terminális Leader fehérjével illetve a picornavírusokra jellemző felépítéssel rendelkezik: 5’ UTR-L-P1(VP0-3-1)-P2(2A-B-C)-P3(3A-B-CD)-3’ UTR (1. ábra).
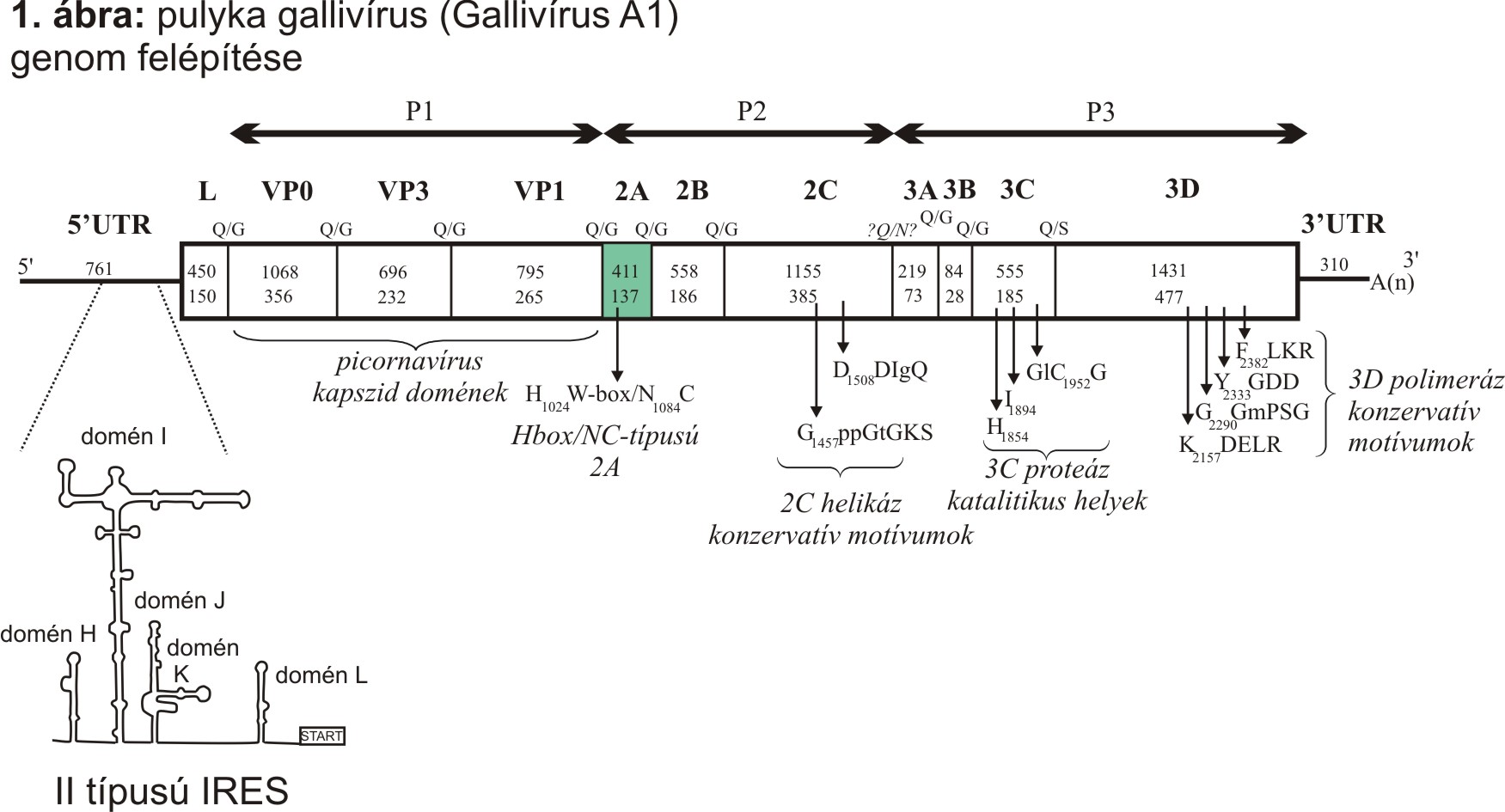
A P1, P2 és P3 régiók 18, 27/32 és 40/45%-os aminosav egyezést mutattak a legközelebbi ismert rokon, Kobuvirus nemzetségbe tartozó sertés kobuvírus 1-el (PoKV-1), illetve a Turdivirus 1-el (jelenleg a Passerivirus nemzetségbe tartozó Passerivirus A1). A vírusgenom egy 150 aa hosszúságú ismeretlen funkciójú Leader (L) proteint, illetve csak egy 137 aa hosszúságú 2A peptidet kódol, amely a Hbox/NC-típusú 2A csoportba tartozik (részletekért lásd: 2014.03.28-ai bejegyzésemet). A pulyka gallivírus távoli rokonságot mutat az énekesmadarakból azonosított passerivírusokkal és oscivírusokkal valamint az emlősök kobuvírusaival (2. ábra). A távoli filogenetikai rokonság többek közt a konzervatív P3 genom régióban, valamint az alacsony szekvencia-egyezési értékek alapján a pulyka galllivírust 2013-ban egy új nemzetségbe, a Gallivirus nemzetségbe sorolták, amelynek prototípus vírustörzse a kutatócsoportunk által azonosított pulyka gallivírus 1, újabb nevén Gallivirus A1 (GenBank ID: JQ691613) lett.
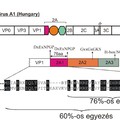
A vírus további érdekessége, hogy ez volt az első ismert madár picornavírus, amelynek 5’ UTR régiója II-es típusú belső riboszóma-kötőhelyet (Internal Ribosomal Entry Site - IRES) tartalmazott (1. ábra). II-es típusú IRES-t addig csak a filogenetikailag távoli Cardio-, Erbo-, Aphtho-, és Parechovirus nemzetségbe tartozó picornavírusok esetében írtak le (részletekért lásd: 2014.05.12-ei bejegyzésemet).
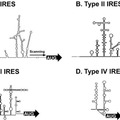
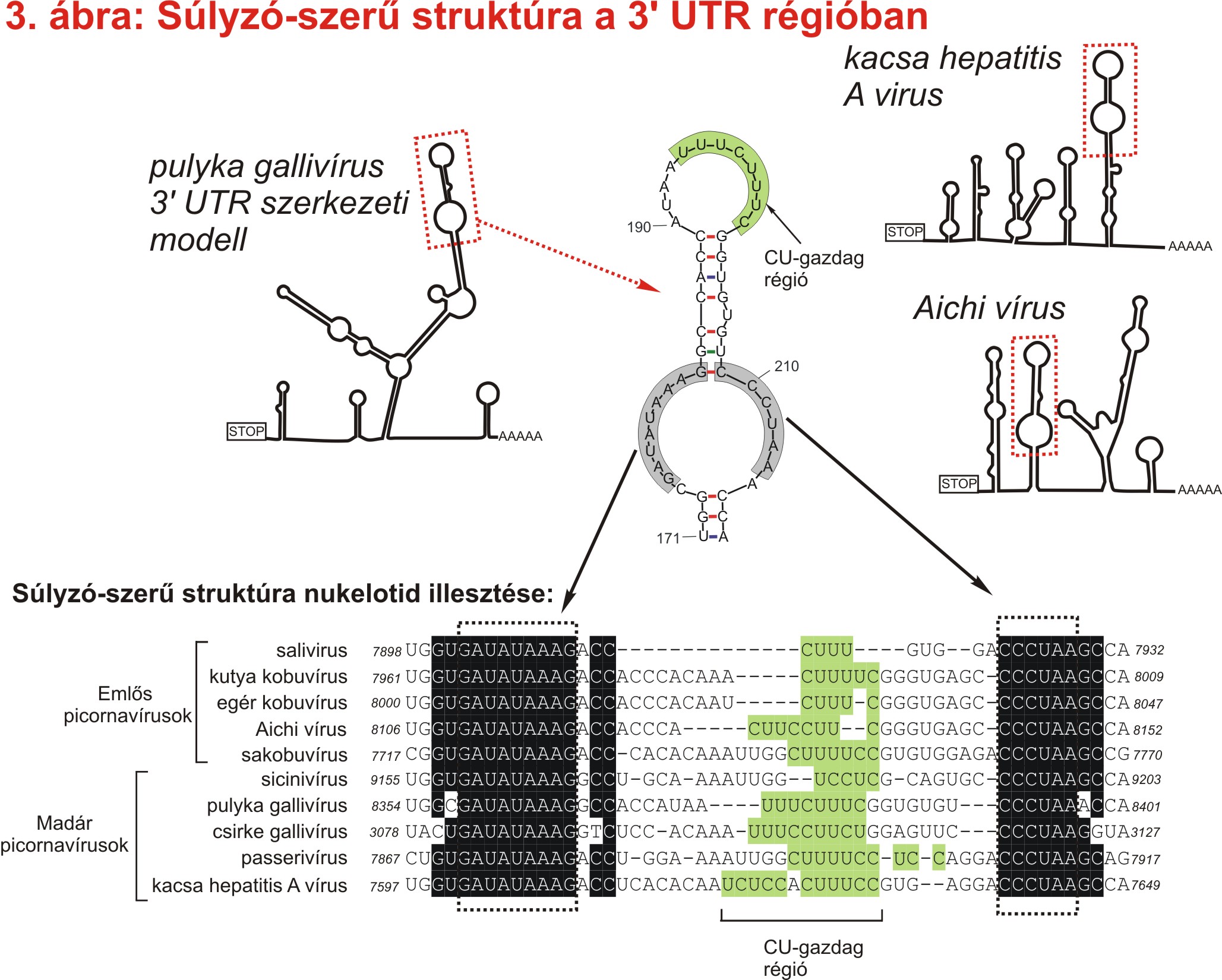
A pulyka gallivírus 310 nt hosszú 3’ UTR régiójában azonosítottunk egy 48 nt hosszúságú szakaszt, ami a másodlagos RNS struktúra modelljeink mindegyikén súlyzó alakot vett fel, és amit alakja miatt súlyzó-szerű (”barbell-like”) stuktúrának neveztük el (3. ábra). A súlyzó-szerű struktúra felső régiója egy rövid pirimidin-gazdag (C és U bázisokból álló) szakaszt tartalmazott (3. ábra).
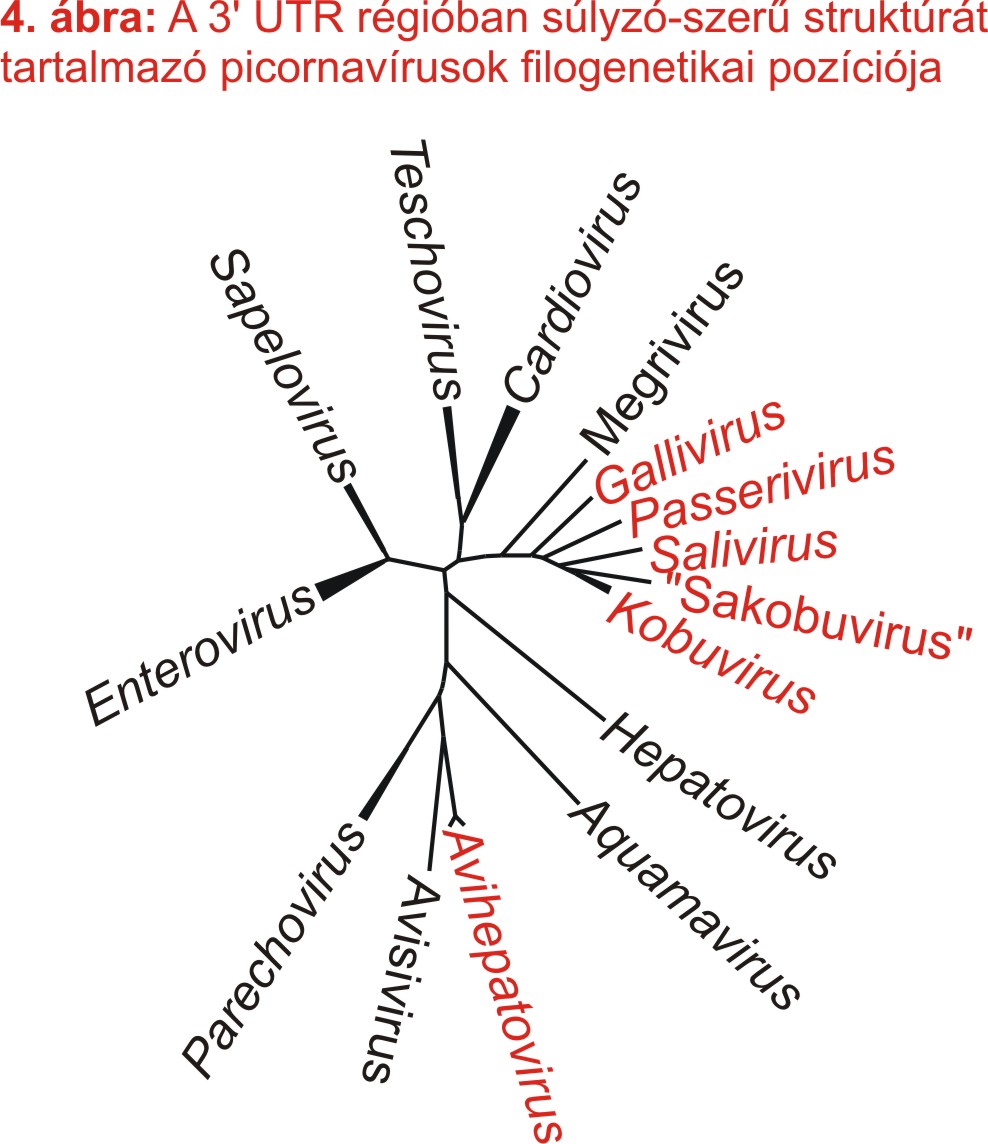
Az összes ismert picornavírusra kiterjedő szekvencia-vizsgálataink kimutatták a súlyzó-szerű struktúra jelenlétét több, a gallivírussal rokon picornavírus 3’ UTR régiójában, valamint a filogenetikailag távoli Avihepatovirus nemzetség vírusaiban is (4. ábra).
A súlyzó-szerű struktúrák a különböző picornavírusok 3’ UTR régiójából készült másodlagos RNS struktúra modelleken a STOP kodontól vírusonként eltérő távolságban helyezkedtek el (3. ábra felső része). A súlyzó-szerű struktúrákat tartalmazó 3’UTR régiók illesztése a felső hurokban található rövid pirimidin-gazdag régió, és a másodlagos RNS struktúra modelleken mindig az alsó hurok részét ábraező, 9+6 nukleotidból álló szekvencia-szinten is konzervatív szakasz jelenlétét mutatta ki (3. ábra). A súlyzó-szerű struktúrák jelenléte filogenetikailag távoli picornavírusok 3’ UTR régióiban, valamint e struktúrák eltérő relatív pozíciója együttesen a súlyzó-szerű struktúrának, mint mobil genetikai elemnek a funkciójára utalhatnak (Boros és mtsai., 2012).
A pulyka gallivírus 1 teljes genomjának leírását követően idén a csirke gallivírus teljes genom szekvenciája is meghatározásra került. A csirke gallivírus szintén II-es típusú IRES-t és a 3’ UTR-jében súlyzó-szerű struktúrát is tartalmaz (Lau és mtsai., 2014).
Referenciák
Farkas, T., Fey, B., Hargitt 3rd, E., Parcells, M., Ladman, B., Murgia, M., Saif, Y., 2012. Molecular detection of novel picornaviruses in chickens and turkeys. Virus Genes 44, 262-272.
Boros, Á., Nemes, C., Pankovics, P., Kapusinszky, B., Delwart, E., Reuter, G., 2012. Identification and complete genome characterization of a novel picornavirus in turkey (Meleagris gallopavo). J. Gen. Virol. 93, 2171–2182.
Lau, S.K., Woo, P.C., Yip, C.C., Li, K.S., Fan, R.Y., Bai, R., Huang, Y., Chan, K.H. and Yuen, K.Y. (2014). Chickens host diverse picornaviruses originated from potential interspecies transmission with recombination. J Gen Virol. 2014 Jun 6. pii: vir.0.066597-0. doi: 10.1099/vir.0.066597-0. [Epub ahead of print] PubMed PMID: 24906980.