Rendhagyó madár picornavírusok bemutatása 1. rész
2014.07.29. 14:46
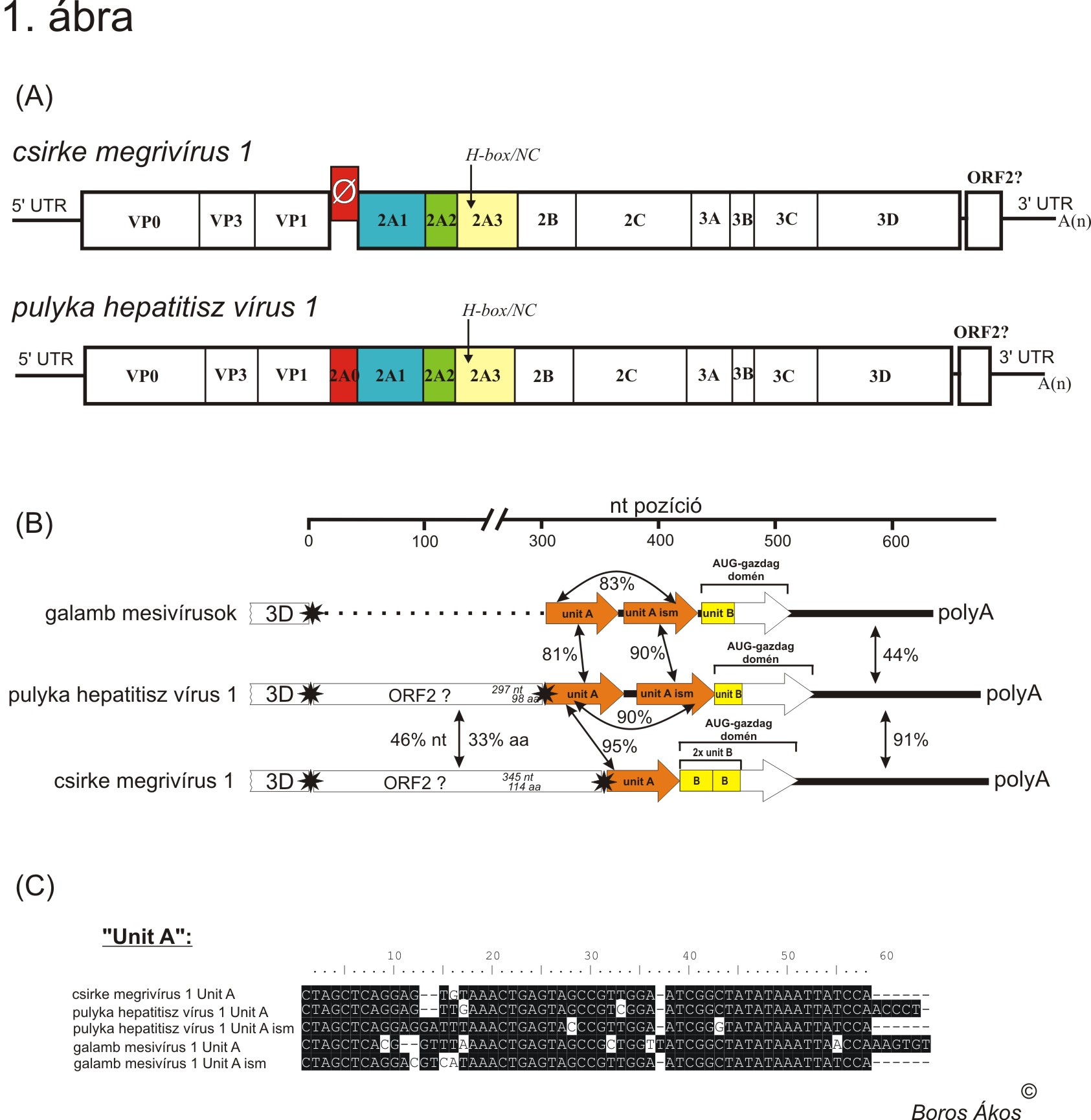
A Megrivírus nemzetség jelenleg 4 fajból áll. A nemzetség prototípus törzsét már 1959-ben izolálták hepatitiszes tüneteket mutató háziasított pulykákból (Meleagris gallopavo f. domestica). Az izolált vírus törzset pulyka hepatitisz vírus 1-esnek (Turkey hepatitis virus 1 / THV-1) nevezték el (Snoeyenbos és mtsai., 1959). A vírus fiziko-kémiai tulajdonságai régóta ismertek, azonban RNS örökítőanyagának kódoló szekvenciáját csak 2011-ben sikerült meghatározni (Honkavuori és mtsai., 2011), az első teljes THV-1 szekvencia pedig csak 2014-ben került meghatározásra (Boros és mtsai., 2014). A THV-1 kódoló szakaszának genetikai vizsgálata kimutatta egy 1800 nukleotid hosszú, ismeretlen fehérjét/fehérjéket kódoló szakasz jelenlétét a genom VP1 és 2B régiója között, amely genom szakasz megfeletethető a picornavírusok 2A régiójának. A szekvencia analízis kimutatta egy már ismert 2A fehérje, az úgynevezett Hbox/NC típusú 2A (erről a 2A típusról részletesen olvashatsz korábbi bejegyzéseimben) jelenlétét ezen a szakaszon. Hasítási helyek elemzése további két potenciális vágási helyet azonosított, ami arra utalt, hogy a THV-1 RNS genomja legalább 3 különböző 2A fehérjét kódol (Honkavuori és mtsai., 2011). Korábban nem volt ismert hasonló picornavírus, amely ilyen hosszú 2A régióval rendelkezett volna. Virális metagenomikai és RT-PCR módszerek felhasználásával egy filogenetikailag és nukleotid-szekvenciában a THV-1-hez közeli rokon picornavírust sikerült izolálni csirkékből (Gallus gallus domesticus) (Boros és mtsai., 2014). A filogenetikai és szekvencia hasonlóság alapján a picornavírus csirke megrivírus 1 néven Megrivírus nemzetségbe sorolható. A csirke megrivírus RNS genomja a THV-1-hez hasonlóan tartalmazott egy hosszú 2A régiót, ami azonban 231 nukleotiddal rövidebbnek, mint a THV-1 azonos szakasza. A 2A régió nukleotid szekvenciáinak illesztése egy 231 nukleotid hosszú inzerciót mutatott ki a THV-1-nél, amely hiányzott a csirke megrivírus genomjából. Az inzerció további vizsgálata egy eddig még nem azonosított, potenciális hasítási hely jelenlétére utalt a THV-1 esetében. Ezek az adatok arra utalnak, hogy a THV-1 összesen 4 különböző 2A fehérjét (2A0-2A1-2A2-2A3) kódol, amelyek közül egy (2A0) valószínűleg kivágódott a csirke megrivírus evolúciós fejlődése során, amely így csak három 2A fehérjét kódol (1. kép A része). Eddigi ismereteink szerint nem ismerünk olyan picornavírust, amely ilyen nagyszámú 2A fehérjét kódolna (Boros és mtsai., 2014).Utóbbi állítás a cikk megjelenése óta hamisnak bizonyult, ugyanis leírtak egy kacsa picornavírust (”Aalivirus”) amely összesen 6 (!) 2A fehérjét kódol (Wang és mtsai., 2014).
További THV-1 rokon vírusokat is leírtak galambokból (Mesivírus 1 és 2). A mesivírusok ”különlegessége”, hogy 2A régiójuk ”csak” két különböző 2A fehérjét kódol (Phan és mtsai., 2013).
A THV-1 és a csirke megrivírusok RNS genomjának másik érdekessége a rendkívül hosszú (>600 nukleotid) 3’ nem kódoló szakasz (3’ UnTranslated Region / 3’ UTR). Szekvencia vizsgálatok egy potenciális második nyílt leolvasási keret (Open Reading Frame / ORF) jelenlétét mutatták ki közvetlenül a virális poliprotein-kódoló régió STOP kodonja után. A potenciális második ORF egy 98 (THV-1), illetve egy 114 (csirke megrivírusok) aminosav hosszú fehérjét kódolhat (Farkas és mtsai., 2012; Boros és mtsai., 2014). Az ORF2 fehérjéi csak kismértékű szekvencia-egyezést mutatnak. A második ORF teljesen hiányzik a galamb mesivírusok genomjából (1. kép B része). Ez a genom-felépítés egyedülálló az ismert picornavírusok között.
A második ORF utáni szakasz, a tényleges 3’ UTR komplex felépítéssel rendelkezik: közvetlen a második ORF után következik egy konzervatív szekvencia szakasz (Unit A), amely a THV-1-nél és a galamb mesivírusoknál ismétlődik, míg a csirke megrivírusoknál csak egy példányban van jelen (1. kép B és C része). A Unit A után egy úgynevezett AUG (Adenin, Uracil és Guanin) gazdag régió következik, amely szintén tartalmaz egy konzervatív szekvencia-szakaszt (Unit B). A Unit B egy példányban van jelen a THV-1-nél és a galamb mesivírusoknál, azonban ismétlődik a csirke megrivírusoknál (1. kép B része) (Boros és mtsai., 2014). Hasonló 3’ UTR felépítést még nem írtak le korábban.
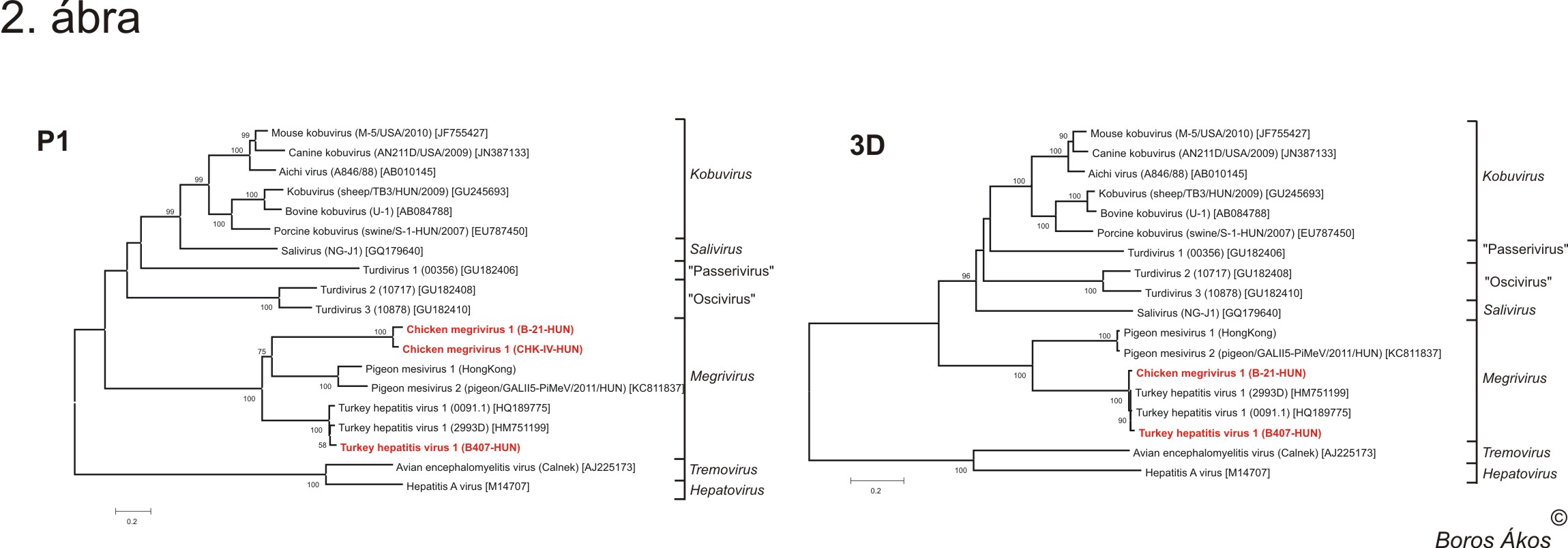
Ha megvizsgáljuk a THV1, a galamb mesivírusok és a csirke megrivírusok filogenetikai pozícióját a kapszid fehérjéket kódoló szakaszon (P1) és a konzervatív 3D RdRp (RNS-függő RNS polymeráz) kódoló szakaszon, akkor a csirke megrivírusokat különböző pozícioban találjuk a két fán (2. kép). A P1 régióban jól látható az elkülönülés a THV-1-től és a galamb mesivírusoktól, míg a 3D f fán a THV-1 és a csirke megrivírusok egy kompakt csoportot alkotnak (2. kép). A filogenetikai pozíciók különbsége a csirke megrivírusok (vagy a THV-1) evolúciós fejlődésében legalább egy, a 2A régióban bekövetkező rekombinációs eseményre utalnak. A feltételezett rekombinációs esemény két, nukleotid szekvenciában távoli (legalább külön fajba tartozó) vírus között jöhetett létre (Boros és mtsai., 2014). A fajok közötti rekombináció nyomai a jelenleg ismert picornavírusok között nagyon ritkák.
A negyedik megrivírus fajt nemrég azonosították kacsákból. A kacsa megrivírus RNS genomja a csirke megrivírusokhoz hasonlóan három különböző 2A fehérjét kódol. Érdekessége, hogy a szintén hosszú (>600 nukleotid) 3’ UTR-je nem mutat hasonlóságot a többi három megrívírussal, nem tartalmaz sem Unit A, sem Unit B szekvenciákat, sem AUG-gazdag régiót. Emellett a második ORF sem kimutatható. A kacsa megrivírus 3’ UTR szekvenciája inkább a kacsa hepatitisz A (Duck hepatitis A / DHAV) 3’ UTR-jével mutat rokonságot (Liao et al., 2014).
Referenciák
Boros, Á., Pankovics, P., Knowles, N.J., Nemes, C., Delwart, E., Reuter, G., 2014.Comparative complete genome analysis of chicken and turkey megriviruses (family Picornaviridae): long 3’ untranslated regions with a potential second open reading frame and evidence for possible recombination. J. Virol. 88, 6434-6443.
Farkas, T., Fey, B., Hargitt 3rd, E., Parcells, M., Ladman, B., Murgia, M., Saif, Y., 2012. Molecular detection of novel picornaviruses in chickens and turkeys. Virus Genes 44, 262-272.
Liao, Q., Zheng, L., Yuan, Y., Shi, J., Zhang, D., 2014. Genomic characterization of a novel picornavirus in Pekin ducks, Vet. Mic. http://dx.doi.org/10.1016/j.vetmic.2014.05.002
Phan, T.G., Vo, N.P., Boros, Á., Pankovics, P., Reuter, G., Li, O.T.W., Wang, C., Deng, X., Poon, L.L.M., Delwart, E., 2013. The viruses of wild pigeon droppings. PLoS ONE 8, e72787.
Wang, X., Liu, N., Wang, F., Ning, K., Li, Y., Zhang, D., 2014.Genetic characterization of a novel duck-origin picornavirus with six 2A proteins. J. Gen. Virol. 95, 1289-1296.
Honkavuori KS, Shivaprasad HL, Briese T, Street C, Hirschberg DL, Hutchison SK, Lipkin WI.2011. Novel picornavirus in turkey poults with hepatitis, California, USA. Emerg. Infect. Dis. 17:480–487.
Snoeyenbos GH, Bosch HI, Sevoian M. 1959. An infectious agent producing hepatitis in turkeys. Avian Diseases. 3:377-388.