Az első madár kobuvírus leírása - szalakóta kobuvírus 1
2015.01.30. 12:19
A Picornaviridae víruscsalád Kobuvírus nemzetségének tagjait számos emlősből kimutatták már (Reuter és mtsai., 2011). A publikus szekvencia-adatbázisokban elérhetőek az emberi (Aichi vírus), kutya, macska, egér, szarvasmarha, sertés és kecske kobuvírusok teljes szekvenciái. Emellett részleges genom szakaszok ismertek egyes denevér és patkány kobuvírusokból is. Az azonosított kobuvírusok 3 hivatalosan elfogadott (Aichivirus A-C) és egy javasolt (”Aichivirus D”) fajba sorolhatóak. Az ismert kobuvírusok mindegyike nagyon hasonló genom-felépítéssel (L-3-3-4) rendelkezik (Reuter és mtsai., 2011). A P1 genom régió előtt egy különböző hosszúságú Leader protein található, amelynek funkciója a nemzetség legtöbb tagjában ismeretlen, csak a humán Aichi vírusok Leader fehérjéjéről tudjuk, hogy fontos szerepet játszik a vírus replikációjának szabályozásában (Sasaki et al., 2003). A P1 strukturális régió három kapszid fehérjét (VP0, VP3 és VP1) kódol. A P2 és P3 nem strukturális genom régiók összesen hét fehérjét kódolnak köztük egy Hbox-NC-típusú 2A-t. A kobuvírusok legtöbbjének 5’ UTR-je V. típusú IRES-t tartalamaz. Ez alól kivételt képeznek a sertés kobuvírusok (Aichivirus C faj tagjai), amelyeknek IV-es IRES-ük van (Reuter és mtsai., 2011). Az Aichivirus A faj összes tagjának, égy a humán Aichi vírusok, valamint a kutya-, macska- és egér kobuvírusok 3’ UTR régiója tartalmazza a gallivírusoknál leírt ”súlyzó-szerű” struktúrát (részletesen lásd előző bejegyzésemet).
A kobuvírusokat még nem mutattak ki madarakból, ezért megvizsgáltuk a kobuvírusok jelenlétét RT-PCR módszerrel szalakótákban (Coracias garrulus). Az alkalmazott UNIV-Kobu-R és F általános kubuvírus szűrő primer-pár segítségével a rendelkezésünkre álló 18 kloáka minta közül háromból (17%-os pozitivitás) sikerült kobuvírust kimutatni. Az egyik kiválasztott mintában (SZAL6) található kobuvírus (SZAL6-KoV/2011/HUN) teljes genom szekvenciája (Azonosító szám: KJ934637) meghatározásra került (Pankovics és mtsai., 2015). A szalakóta kobuvírus 1 (European roller kobuvirus 1 - ERKV-1) genomja 8251 nukleotid (nt) hosszúságú, és a bevezetésben említett általános kobuvírus genom-felépítést (L-3-3-4) követi (1. kép).
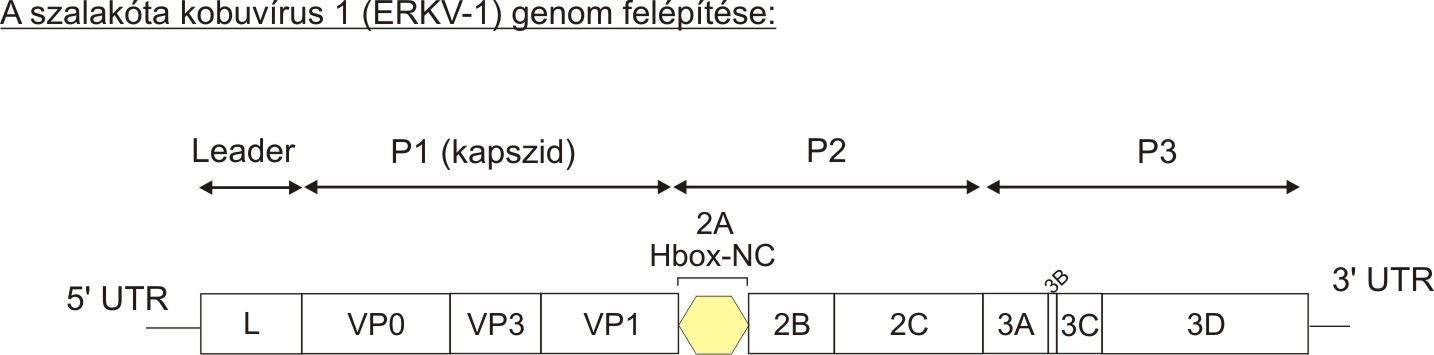
Leader proteinjének funkciója ismeretlen, 2A fehérjéje pedig a többi kobuvírus 2A-hoz hasonlóan Hbox-NC-típusú 2A fehérjék csoportjába tartozik. Törzsfejlődéstani és szekvencia-hasonlósági szempontból az ERKV-1 legközelebbi rokonságot az Aichivirus A faj tagjaival, ezen belül is a humán Aichi vírussal (72%-73%-81% aminosav egyezés a P1-P2-P3 régiókban) mutatta (2. kép).
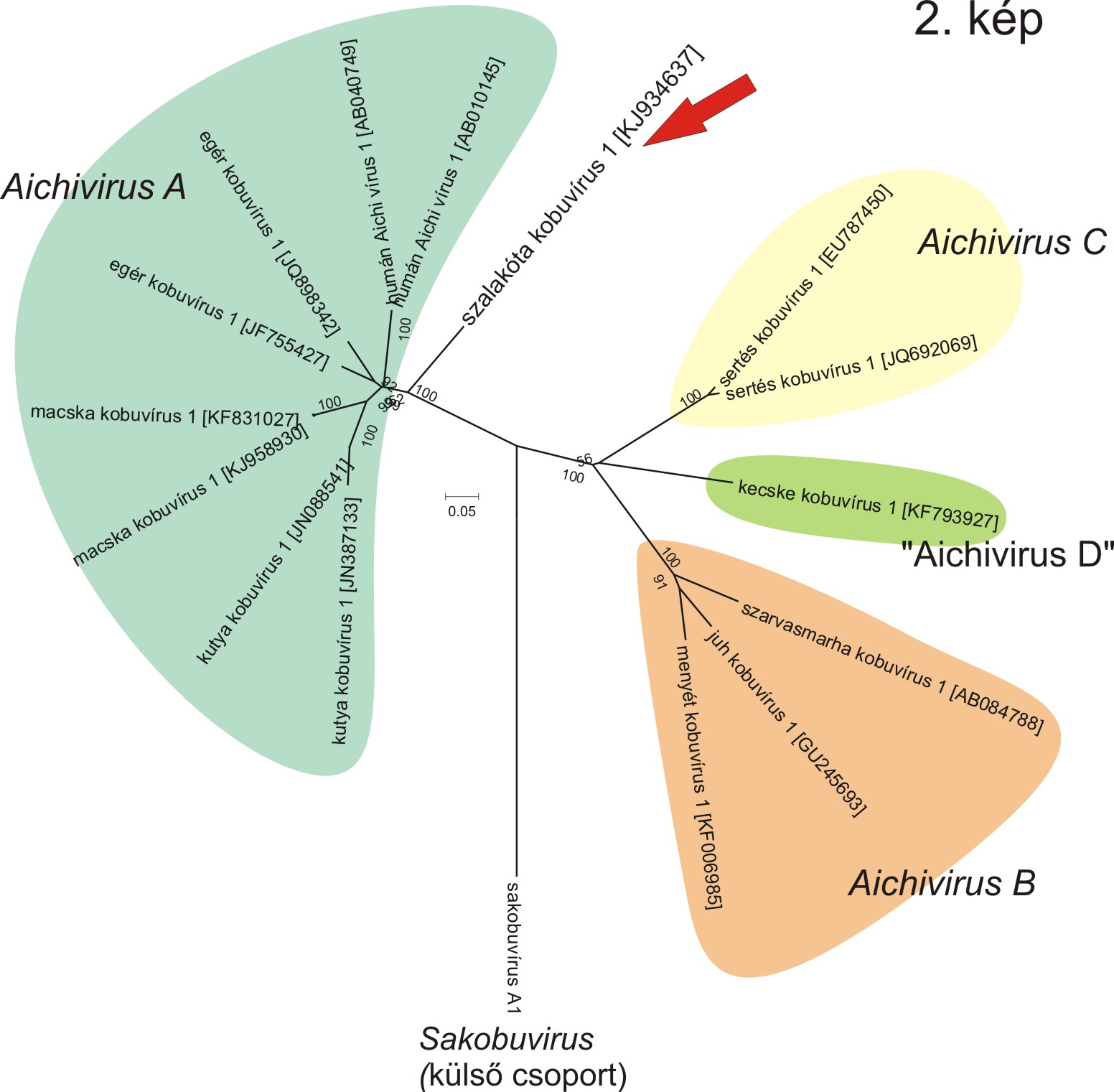
Mindezekből arra következtethetünk, hogy a szalakóta kobuvírus 1 és az Aichivirus A faj kobuvírusai azonos őstől származhatnak. A közös származásra az ERKV-1 és Aichivirus A faj tagjainak azonos, V. típusú IRES-e is utalhat. Érdekességként jelezném meg, hogy a vizsgált vírusunk 3’ UTR régiója nem tartalmazza az összes Aichivirus A faj tagjára jellemző ”súlyzó-szerű” struktúrát (Pankovics és mtsai., 2015). A filogenetikai és szekvencia-vizsgálataink eredményei nem teszik lehetővé az ERKV-1 pontos, faj-szintű besorolását. A vizsgált vírus vagy egy új kobuvírus faj (”Aichivirus E”) első tagja, vagy az Aichivirus A fajba tartozó szekvencia.
Referenciák
Sasaki J, Nagashima S, Taniguchi K. Aichi Virus Leader Protein Is Involved in Viral RNA Replication and Encapsidation. J Virol. 2003 October; 77(20): 10799–10807.
Reuter G, Boros Á , Pankovics P (2011) Kobuviruses—a comprehensive review. Rev Med Virol 21:32–41.
Pankovics P., Boros Á., Kiss T. and Reuter G. (2015). Identification and complete genome analysis of kobuvirus in faecal samples of European roller (Coracias garrulus): for the first time in bird. Arch. Virol. 60:345–351.